CellViT:基于视觉Transformer的精确细胞分割与分类方法
CellViT:基于视觉Transformer的精确细胞分割与分类方法
CellViT是由德国埃森大学医院人工智能医学研究所(IKIM)开发的一种新型深度学习方法,用于对数字化组织样本中的细胞核进行自动化实例分割。这项研究成果发表在Medical Image Analysis期刊上,论文题为《CellViT: Vision Transformers for precise cell segmentation and classification》。
研究背景与意义
在血细胞和嗜酸性组织(H&E)染色的组织图像中进行细胞核检测和分割是一项重要的临床任务,对于许多应用来说都至关重要。然而,由于细胞核在染色和大小上的变异性,以及细胞核边界重叠和聚集等因素,这项任务仍然具有挑战性。虽然卷积神经网络已被广泛应用于该任务,但研究人员开始探索基于Transformer的网络在这一领域的潜力。
CellViT方法简介
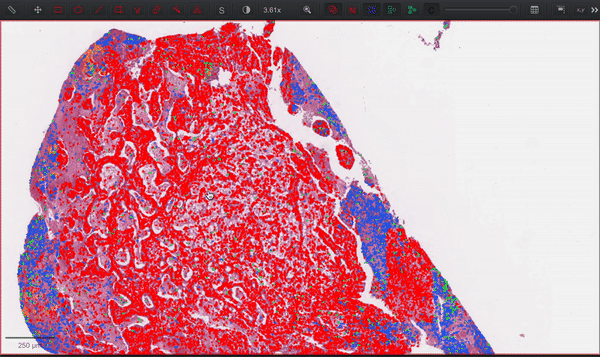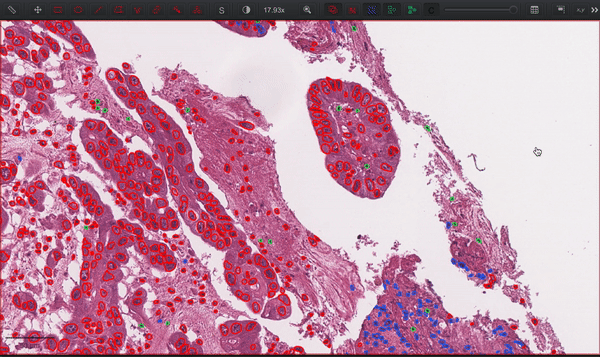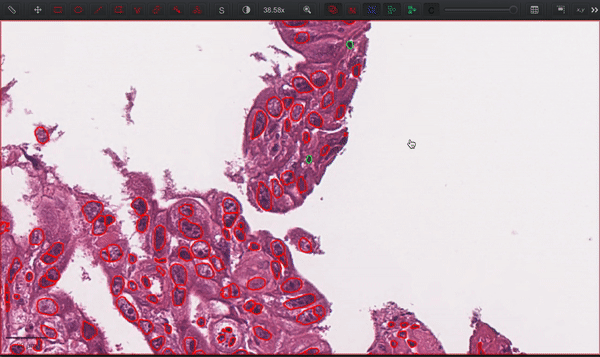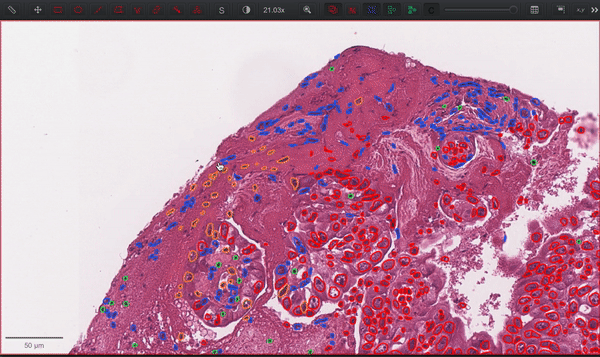
CellViT是一种基于视觉Transformer的深度学习架构,用于对数字化组织样本中的细胞核进行自动化实例分割。该方法在PanNuke数据集上进行了训练和评估,这是一个具有挑战性的细胞核实例分割数据集,包含了19种组织类型中近20万个带注释的细胞核,分为5个临床重要类别。
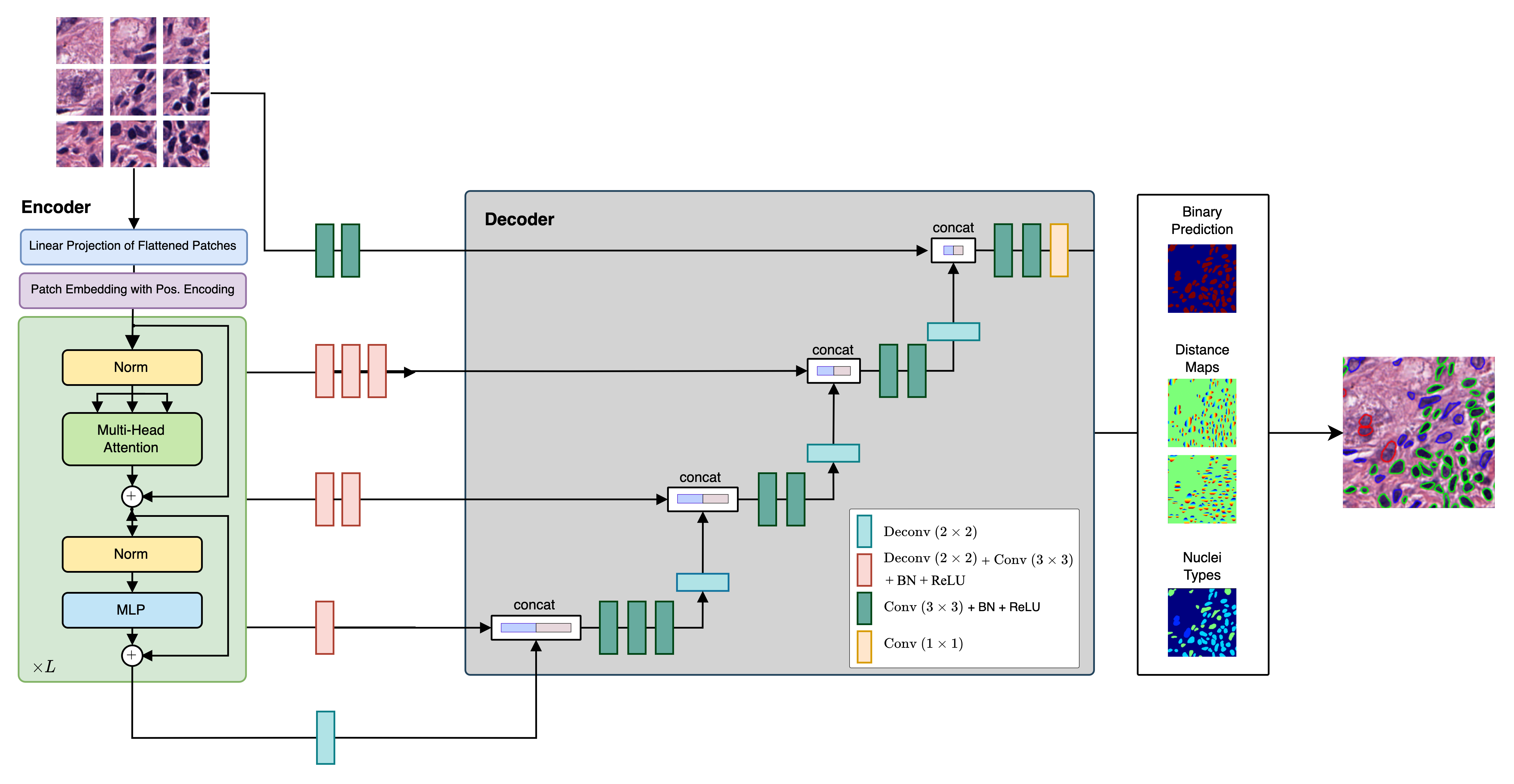
CellViT的主要特点包括:
-
视觉Transformer编码器:项目采用了预训练的视觉Transformer(ViT)编码器,这种编码器在各种计算机视觉任务中都表现出色。这一选择提高了CellViT的分割性能。
-
U-Net架构:CellViT采用了U-Net形状的编码器-解码器网络结构,可以高效准确地进行细胞核实例分割。该网络架构有助于提取高层和低层特征,从而改善分割结果。
-
加权采样策略:为了提高CellViT的性能,研究人员引入了一种新的加权采样策略。这一策略改善了对具有挑�战性的细胞核实例的表示,从而得到更准确的分割结果。
-
千兆像素WSI快速推理:该框架通过使用1024×1024像素的大型推理patch尺寸(相比传统的256像素patch),实现了快速推理。这种方法能够高效分析千兆像素级别的全扫描图像(WSI),并生成可本地化的深度特征,为下游任务提供潜在价值。
实验结果与性能
CellViT在PanNuke数据集上实现了最先进的细胞核检测和实例分割性能:
- 平均全景质量(PQ):0.51
- F1检测分数:0.83
研究人员通过利用最近发布的Segment Anything Model(SAM)和在1.04亿个组织学图像patch上预训练的ViT编码器,展示了大规模领域内和领域外预训练的视觉Transformer的优越性。
开源与应用
CellViT项目的代码已在GitHub上开源(https://github.com/TIO-IKIM/CellViT),研究人员可以自由使用和改进这一方法。项目提供了详细的安装指南、使用说明和预处理流程,方便其他研究人员复现结果和在自己的数据集上应用CellViT。
此外,研究团队还提供了预训练模型的下载链接,包括:
- CellViT-SAM-H
- CellViT-256
- CellViT-SAM-H-x20
- CellViT-256-x20
这些模型使用了90%的所有折叠数据进行训练,采用了论文中描述的设置。
未来展望
CellViT项目团队目前正在优化推理速度,相关代码将在未来几周内更新。此外,他们计划在未来的版本中提供一个Docker镜像,其中包含所有必要的依赖项和预安装的配置。这个Docker镜像将确保可重复性并简化设置过程,允许轻松安装和使用该项目。
总的来说,CellViT为细胞核实例分割任务提供了一种强大而灵活的解决方案。通过结合视觉Transformer的优势和创新的网络架构设计,CellViT在challenging的PanNuke数据集上取得了优异的性能。这项研究不仅推动了医学图像分析领域的发展,也为其他领域的实例分割任务提供了新的思路和方法。随着项目的持续改进和社区的参与,CellViT有望在未来发挥更大的作用,为医学研究和临床实践做出重要贡献。
编辑推荐精选

AEE
AI Excel全自动制表工具
AEE 在线 AI 全自动 Excel 编辑器,提供智能录入、自动公式、数据整理、图表生成等功能,高效处理 Excel 任务,提升办公效率。支持自动高亮数据、批量计算、不规则数据录入,适用于企业、教育、金融等多场景。


UI-TARS-desktop
基于 UI-TARS 视觉语言模型的桌面应用,可通过自然语言控制计算机进行多模态操作。
UI-TARS-desktop 是一款功能强大的桌面应用,基于 UI-TARS(视觉语言模型)构建。它具备自然语言控制、截图与视觉识别、精确的鼠标键盘控制等功能,支持跨平台使用(Windows/MacOS),能提供实时反馈和状态显示,且数据完全本地处理,保障隐私安全。该应用集成了多种大语言模型和搜索方式,还可进行文件系统操作。适用于需要智能交互和自动化任务的场景,如信息检索、文件管理等。其提供了详细的文档,包括快速启动、部署、贡献指南和 SDK 使用说明等,方便开发者使用和扩展。


Wan2.1
开源且先进的大规模视频生成模型项目
Wan2.1 是一个开源且先进的大规模视频生成模型项目,支持文本到图像、文本到视频、图像到视频等多种生成任务。它具备丰富的配置选项,可调整分辨率、扩散步数等参数,还能对提示词进行增强。使用了多种先进技术和�工具,在视频和图像生成领域具有广泛应用前景,适合研究人员和开发者使用。


爱图表
全流程 AI 驱动的数据可视化工具,助力用户轻松创作高颜值图表
爱图表(aitubiao.com)就是AI图表,是由镝数科技推出的一款创新型智能数据可视化平台,专注于为用户提供便捷的图表生成、数据分析和报告撰写服务。爱图表是中国首个在图表场景接入DeepSeek的产品。通过接入前沿的DeepSeek系列AI模型,爱图表结合强大的数据处理能力与智能化功能,致力于帮助职场人士高效处理和表达数据,提升工作效率和报告质量。


Qwen2.5-VL
一款强大的视觉语言模型,支持图像和视频输入
Qwen2.5-VL 是一款强大的视觉语言模型,支持图像和视频输入,可用于多种场景,如商品特点总结、图像文字识别等。项目提供了 OpenAI API 服务、Web UI 示例等部署方式,还包含了视觉处理工具,有助于开发者快速集成和使用,提升工作效率。


HunyuanVideo
HunyuanVideo 是一个可基于文本生成高质量图像和视频的项目。
HunyuanVideo 是一个专注于文本到图像及视频生成的项目。它具备强大的视频生成能力,支持多种分辨率和视频长度选择,能根据用户输入的文本生成逼真的图像和视频。使用先进的技术架构和算法,可灵活调整生成参数,满足不同场景的需求,是文本生成图像视频领域的优质工具。


WebUI for Browser Use
一个基于 Gradio 构建的 WebUI,支持与浏览器智能体进行便捷交互。
WebUI for Browser Use 是一个强大的项目�,它集成了多种大型语言模型,支持自定义浏览器使用,具备持久化浏览器会话等功能。用户可以通过简洁友好的界面轻松控制浏览器智能体完成各类任务,无论是数据提取、网页导航还是表单填写等操作都能高效实现,有利于提高工作效率和获取信息的便捷性。该项目适合开发者、研究人员以及需要自动化浏览器操作的人群使用,在 SEO 优化方面,其关键词涵盖浏览器使用、WebUI、大型语言模型集成等,有助于提高网页在搜索引擎中的曝光度。


xiaozhi-esp32
基于 ESP32 的小智 AI 开发项目,支持多种网络连接与协议,实现语音交互等功能。
xiaozhi-esp32 是一个极具创新性的基于 ESP32 的开发项目,专注于人工智能语音交互领域。项目涵盖了丰富的功能,如网络连接、OTA 升级、设备激活等,同时支持多种��语言。无论是开发爱好者还是专业开发者,都能借助该项目快速搭建起高效的 AI 语音交互系统,为智能设备开发提供强大助力。


olmocr
一个用于 OCR 的项目,支持多种模型和服务器进行 PDF 到 Markdown 的转换,并提供测试和报告功能。
olmocr 是一个专注于光学字符识别(OCR)的 Python 项目,由 Allen Institute for Artificial Intelligence 开发。它支持多种模型和服务器,如 vllm、sglang、OpenAI 等,可将 PDF 文件的页面转换为 Markdown 格式。项目还提供了测试框架和 HTML 报告生成功能,方便用户对 OCR 结果进行评估和分析。适用于科研、文档处理等领域,有助于提高工作效率和准确性。


飞书多维表格
飞书多维表格 ×DeepSeek R1 满血版
飞书多维表格联合 DeepSeek R1 模型,提供 AI 自动化解决方案,支持批量写作、数据分析、跨模态处理等功能,适用于电商、短视频、影视创作等场景,提升企业生产力与创作效率。关键词:飞书多维表格、DeepSeek R1、AI 自动化、批量处理、企业协同工具。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号







