单细胞转换器模型的发展与应用
引言
近年来,单细胞测序技术的飞速发展为我们提供了前所未有的机会来深入理解细胞异质性和基因调控网络。然而,如何有效分析和利用这些海量的单细胞数据仍然是一个巨大的挑战。在这一背景下,基于transformer架构的深度学习模型因其强大的特征提取和表示学习能力,逐渐成为单细胞数据分析的新兴工具。本文将全面回顾单细胞转换器模型的最新进展,探讨其在生物信息学领域的广泛应用前景。
单细胞转换器模型概述
单细胞转换器模型是一类将transformer架构应用于单细胞数据分析的深度学习模型。这些模型通常采用自监督学习的方式在大规模单细胞数据集上进行预训练,学习细胞和基因的通用表示,然后通过微调或特征提取的方式应用于各种下游任务。
模型架构
大多数单细胞转换器模型采用编码器架构,将细胞或基因作为输入token。例如,scGPT[1]和Geneformer[2]等模型采用了类似BERT的双向编码器架构,而scMulan[3]则使用了解码器架构。一些模型如scFoundation[4]还采用了双编码器设计。在输入表示方面,常见的方法包括值投影、值分类和排序等。
预训练策略
预训练是单细胞转换器模型的关键步骤。常用的预训练任务包括:
- 掩码语言模型(MLM): 预测被掩码的基因表达值,是最常用的预训练任务。
- 对比学习: 学习不同细胞或基因之间的相似性。
- 基因ID预测: 预测基因的身份信息。
- 细胞类型预测: 在有标注数据的情况下进行监督预训练。
许多模型还采用了多任务学习的策略,同时优化多个预训练目标。
数据集和规模
预训练数据集的规模和多样性对模型性能有重要影响。目前规模最大的预训练数据集来自GeneCompass[5],包含了126M个人类和小鼠细胞。其他大规�模数据集还有CELLxGENE[6]和Genecorpus[2]等。这些数据集通常涵盖了多个组织和物种,包括健康和疾病样本。
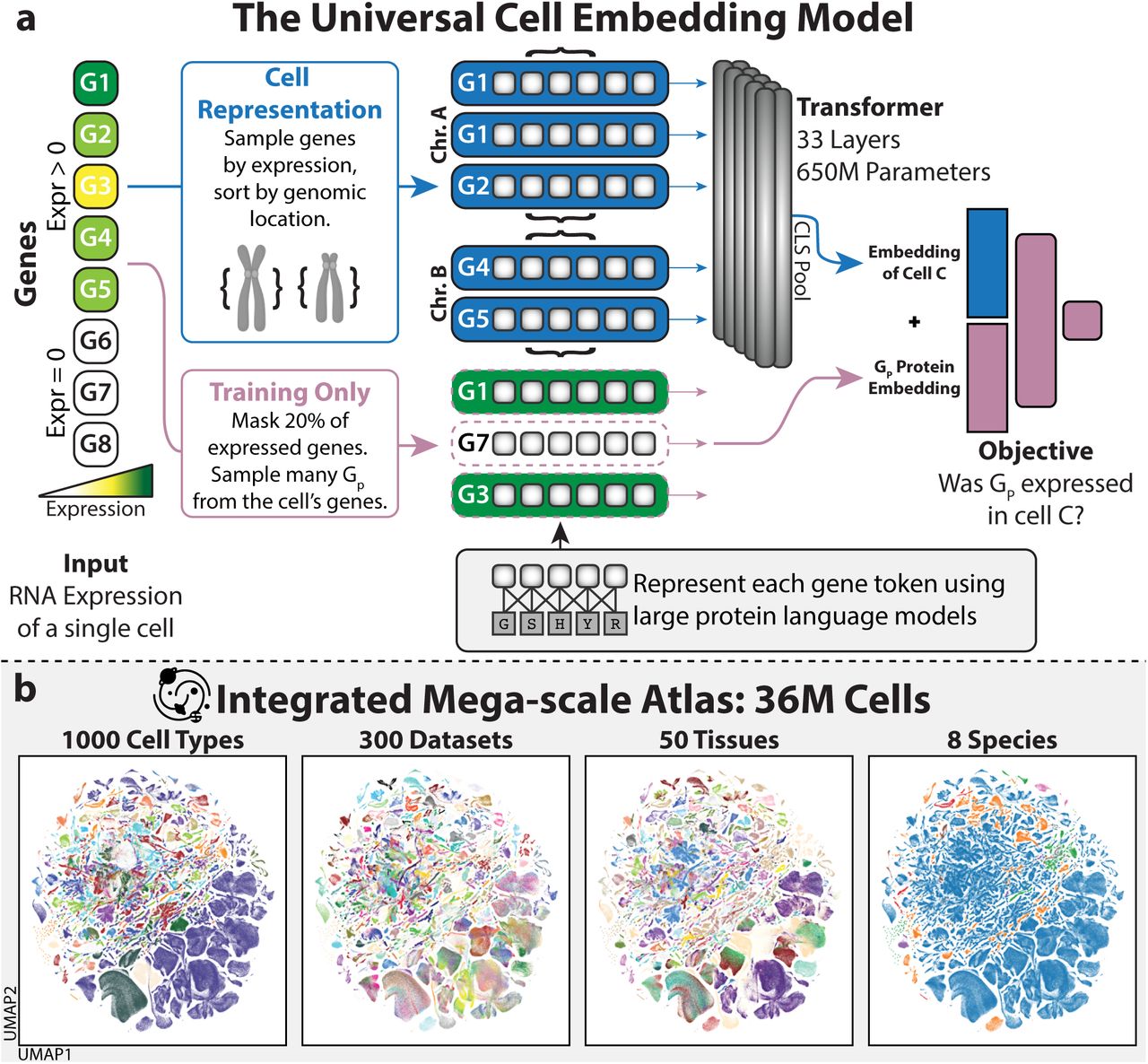
下游应用
单细胞转换器模型在多个生物信息学任务中展现出了强大的性能:
细胞类型注释
细胞类型注释是单细胞数据分析的基础任务。多个模型如scBERT[7]、TOSICA[8]和scGPT[1]等都在这一任务上取得了优异的表现。这些模型能够学习到细胞的通用表示,有效捕捉不同细胞类型的特征。
基因调控网络推断
基因调控网络(GRN)推断是理解基因间相互作用的重要方法。Geneformer[2]、scGPT[1]和GeneCompass[5]等模型通过学习基因的上下文相关表示,为GRN推断提供了新的思路。
细胞轨迹分析
细胞轨迹分析旨在揭示细胞分化和状态转换的动态过程。tGPT[9]等模型通过学习细胞的时序表示,在轨迹推断任务中展现出优势。
跨模态整合
随着多组学测序技术的发展,如何有效整合不同模态的单细胞数据成为一个重要问题。scCLIP[10]和scMoFormer[11]等模型通过联合建模多种组学数据,实现了跨模态的数据整合和预测。
药物响应预测
预测细胞对药物的响应是药物开发的关键步骤。CellLM[12]和scFoundation[4]等模型通过整合基因表达和药物特征,为个性化医疗提供了新的工具。
挑战与展望
尽管单细胞转换器模型取得了显著进展,但仍面临一些挑战:
- 计算资源: 大规模模型的训练和部署需要大量计算资源,如何提高模型效率是一个重要问题。
- 解释性: 提高模型的可解释性,使生物学家能更好理解模型的决策过程。
- 整合先验知识: 如何将已有的生物学知识有效整合到模��型中。
- 泛化能力: 提高模型在不同数据集和任务间的泛化能力。
未来,单细胞转换器模型有望在以下方向取得突破:
- 多模态整合: 开发能同时处理转录组、表观组、蛋白组等多种组学数据的统一模型。
- 时空动态建模: 结合空间转录组学数据,构建细胞的时空动态模型。
- 个性化医疗: 利用大规模单细胞数据训练的模型,为疾病诊断和治疗提供个性化的建议。
- 药物开发: 辅助新药靶点的发现和药物筛选过程。
结论
单细胞转换器模型为单细胞数据分析带来了新的机遇,有望推动生物信息学和精准医疗领域的重大进展。随着模型架构、训练策略和应用范围的不断创新,我们期待这一领域在未来能够产生更多令人兴奋的突破。
参考文献
[1] Cui, H. et al. Nat. Methods (2024). [2] Theodoris, C. V. et al. Nature 620, 161–170 (2023). [3] Bian, Y. et al. RECOMB 2024, 1002-1019 (2024). [4] Hao, Y. et al. Nat. Methods (2024). [5] Yang, K. K. et al. bioRxiv (2023). [6] Rosen, Z. et al. bioRxiv (2023). [7] Yang, F. et al. Nat. Mach. Intell. 4, 1013–1026 (2022). [8] Chen, J. et al. Nat. Commun. 14, 330 (2023). [9] Shen, Z. et al. iScience 26, 106536 (2023). [10] Xiong, L. et al. arXiv:2310.07079 (2023). [11] Tang, J. et al. ACM Conference (2023). [12] Zhao, T. et al. arXiv:2306.04371 (2023).
编辑推荐精选

AEE
AI Excel全自动制表工具
AEE 在线 AI 全自动 Excel 编辑器,提供智能录入、自动公式、数据整理、图表生成等功能,高效处理 Excel 任务,提升办公效率。支持自动高亮数据、批量计算、不规则数据录入,适用于企业、教育、金融等多场景。


UI-TARS-desktop
基于 UI-TARS 视觉语言模型的桌面应用,可通过自然语言控制计算机进行多模态操作。
UI-TARS-desktop 是一款功能强大的桌面应用,基于 UI-TARS(视觉语言模型)构建。它具备自然语言控制、截图与视觉识别、精确的鼠标键盘控制等功能,支持跨平台使用(Windows/MacOS),能提供实时反馈和状态显示,且数据完全本地处理,保障隐私安全。该应用集成了多种大语言模型和搜索方式,还可进行文件系统操作。适用于需要智能交互和自动化任务的场景,如信息检索、文件管理等。其提供了详细的文档,包括快速启动、部署、贡献指南和 SDK 使用说明等,方便开发者使用和扩展。


Wan2.1
开源且先进的大规模视频生成模型项目
Wan2.1 是一个开源且先进的大规模视频生成模型项目,支持文本到图像、文本到视频、图像到视频等多种生成任务。它具备丰富的配置选项,可调整分辨率、扩散步数等参数,还能对提示词进行增强。使用了多种先进技术和工具,在视频和图像生成领域具有广泛应用前景,适合研究人员和开发者使用。


爱图表
全流程 AI 驱动的数据可视化工具,助力用户轻松创作高颜值图表
爱图表(aitubiao.com)就是AI图表,是由镝数科技推出的一款创新型智能数据可视化平台,专注于为用户提供便��捷的图表生成、数据分析和报告撰写服务。爱图表是中国首个在图表场景接入DeepSeek的产品。通过接入前沿的DeepSeek系列AI模型,爱图表结合强大的数据处理能力与智能化功能,致力于帮助职场人士高效处理和表达数据,提升工作效率和报告质量。


Qwen2.5-VL
一款强大的视觉语言模型,支持图像和视频输入
Qwen2.5-VL 是一款强大的视觉语言模型,支持图像和视频输入,可用于多种场景,如商品特点总结、图像文字识别等。项目提供了 OpenAI API 服务、Web UI 示例等部署方式,还包含了视觉处理工具,有助于开发者快速集成和使用,提升工作效率。


HunyuanVideo
HunyuanVideo 是一个可基于文本生成高质量图像和视频的项目。
HunyuanVideo 是一个专注于文本到图像及视频生成的项目。它具备强大的视频生成能力,支持多种分辨率和视频长度选择,能根据用户输入的文本生成逼真的图像和视频。使用先进的技术架构和算法,可灵活调整生成参数,满足不同场景的需求,是文本生成图像视频领域的优质工具。


WebUI for Browser Use
一个基于 Gradio 构建的 WebUI,支持与浏览器智能体进行便捷交互。
WebUI for Browser Use 是一个强大的项目,它集成了多种大型语言模型,支持自定义浏览器使用,具备持久化浏览器会话等功能。用户可以通过简洁友好的界面轻松控制浏览器智能体完成各类任务,无论是数据提取、网页导航还是表单填写等操作都能高效实现,有利于提高工作效率和获取信息的便捷性。该项目适合开发者、研究人员以及需要自动化浏览器操作的人群使用,在 SEO 优化方面,其关键词涵盖浏览器使用、WebUI、大型语言模型集成等,有助于提高网页在搜索引擎中的曝光度。


xiaozhi-esp32
基于 ESP32 的小智 AI 开发项目,支持多种网络连接与协议,实现语音交互等功能。
xiaozhi-esp32 是一个极具创新性的基于 ESP32 的开发项目,专注于人工智能语音交互领域。项目涵盖了丰富的功能,如网络连接、OTA 升级、设备激活等,同时支持多种语言。无论是开发爱好者还是专业开发者,都能借助该项目快速搭建起高效的 AI 语音交互系统,为智能设备开发提供强大助力。


olmocr
一个用于 OCR 的项目,支持多种模型和服务器进行 PDF 到 Markdown 的转换,并提供测试和报告功能。
olmocr 是一个专注于光学字符识别(OCR)的 Python 项目,由 Allen Institute for Artificial Intelligence 开发。它支持多种模型和服务器,如 vllm、sglang、OpenAI 等,可将 PDF 文件的页面转换为 Markdown 格式。项目还提供了测试框架和 HTML 报告生成功能,方便用户对 OCR 结果进行评估和分析。适用于科研、文档处理等领域,有助于提高工作效率和准确性。


飞书多维表格
飞书多维表格 ×DeepSeek R1 满血版
飞书多维表格联合 DeepSeek R1 模型,提供 AI 自动化解决方案,支持批量写作、数据分析、跨模态处理等功能,适用于电商、短视频、影视创作等场景,提升企业生产力与创作效率。关键词:飞书多维表格、DeepSeek R1、AI 自动化、批量处理、企业协同工具。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号







