owmeta简介
owmeta是一个为OpenWorm项目开发的Python数据访问库,旨在整合和简化C. elegans(秀丽隐杆线虫)的解剖学和生理学数据访问。它为研究人员提供了一个统一的接口,用于查询和分析与C. elegans相关的各种异构数据和模型。
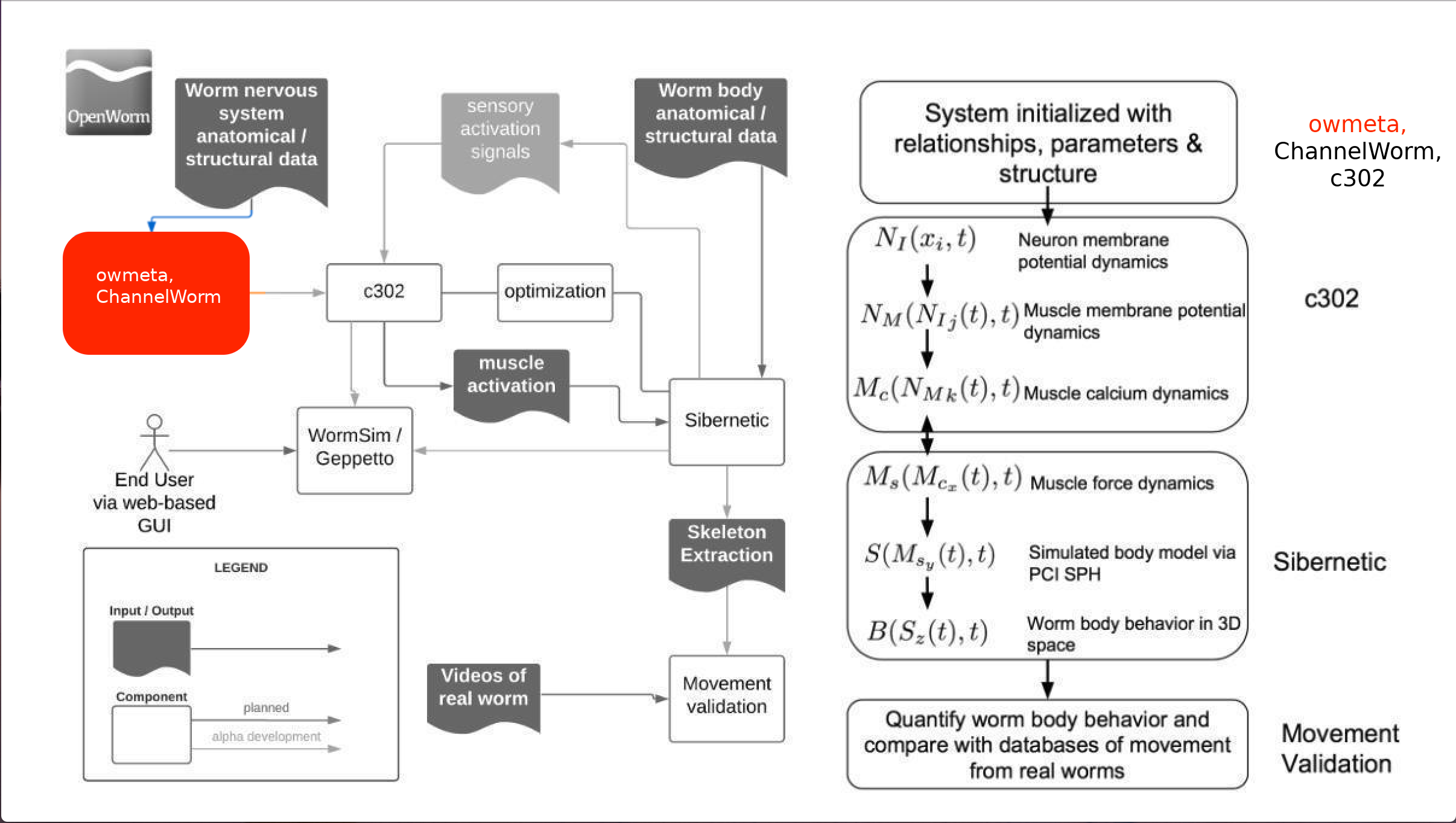
主要特点
-
统一的数据访问层:整合了多种数据表示形式,如NetworkX图、RDF语义图、NeuroML等。
-
简洁的Python API:可以轻松查询C. elegans细胞的各种信息。
-
数据共享功能:支持OpenWorm项目的数据到模型流水线构建。
-
版本控制:保证了数据的一致性和可重复性。
-
证据支持:可以将数据和模型与同行评审文献关联。
技术原理
owmeta通过抽象视图封装了不同的底层数据表示,使用户可以直接操作与蠕虫生物学现实相关的对象,而无需关心底层使用了哪种表示方法。这种设计解决了OpenWorm项目中数据整合和整合的挑战。
主要的数据表示包括:
- NetworkX图:用于表示神经元连接组,方便查询神经元的邻近关系。
- RDF语义图:用于读写神经元的多方面注释,如相关文献、离子通道和神经递质受体等属性。
- NeuroML:用于回答关于模型形态和仿真参数的问题。
- Blender表示:提供完整的3D形状定义,可用于3D空间计算。
owmeta将这些异构的表示整合到一个统一的数据访问层中,使研究人员可以专注于生物学问题,而不是数据表示的技术细节。
使用示例
以下是一些使用owmeta查询C. elegans数据的示例:
# 连接数据库
from owmeta_core.command import OWM
conn = OWM().connect()
# 创建上下文
from owmeta_core.context import Context
ctx = conn(Context)(ident='http://openworm.org/data')
# 获取神经网络表示
from owmeta.worm import Worm
net = ctx.stored(Worm).query().neuron_network()
# 获取特定神经元
from owmeta.neuron import Neuron
aval = ctx.stored(Neuron).query(name='AVAL')
# 获取神经元类型
aval.type.one()
# 输出: 'interneuron'
# 计算从AVAL出发的连接数量
aval.connection.count('pre')
# 输出: 86
# 列出所有已知的受体
sorted(aval.receptors())
# 输出: ['GGR-3', 'GLR-1', ..., 'NPR-4', 'UNC-8']
# 获取所有神经元名称
sorted(net.neuron_names())
# 输出: ['ADAL', 'ADAR', ..., 'VD8', 'VD9']
# 获取所有肌肉
muscles = ctx.stored(Worm).query().muscles()
len(muscles)
# 输出: 158
这些示例展示了owmeta强大的查询能力,可以方便地获取神经元信息、连接关系、受体、肌肉等数据。
证据支持
owmeta还支持将数据和模型与同行评审文献关联,以确保数据的可靠性。例如:
from owmeta.document import Document
from owmeta.evidence import Evidence
# 创建证据上下文
evctx = conn(Context)(ident='http://example.org/evidence/context')
# 创建领域知识上下文
dctx = evctx(Context)(ident='http://example.org/data/context')
# 创建文献引用
doc = evctx(Document)(key="Sulston83", author='Sulston et al.', date='1983')
# 创建证据
e = evctx(Evidence)(key="Sulston83", reference=doc)
# 将证据与数据关联
avdl = dctx(Neuron)(name="AVDL")
avdl.lineageName("AB alaaapalr")
e.supports(dctx.rdf_object)
# 保存上下文
with conn.transaction_manager:
dctx.save_context()
evctx.save_context()
这种机制确保了owmeta中的生理数据具有科学依据,提高了数据的可信度。
与ChannelWorm2的关系
ChannelWorm2是OpenWorm的一个子项目,专门用于存储离子通道模型。虽然目前owmeta也包含了大量的C. elegans生理信息,但未来ChannelWorm2将作为owmeta的"消费者"。owmeta数据库将存储物理模型、数字化图表以及将这些模型转换为可枚举格式的代码。
最终,owmeta的核心将专注于作为存储元数据和来源信息(如与仿真相关的参数和轨迹)的数据框架,并将作为独立功能分离出来。
数据版本控制
owmeta采用了数据版本控制的方法,以确保库的稳定性和数据的一致性。这种方法解决了动态数据暴露可能导致的问题,如数据集变化导致查询结果不一致。
具体措施包括:
-
版本保证:owmeta库的版本保证用户获得特定版本的底层数据。
-
单元测试:使用单元测试确保数据的基本完整性检查得以维护。
-
回归测试:在每个新版本发布前,维护人员会进行适当的回归测试,以保证稳定性。
这种版本控制方法为其他人重用owmeta提供了一个稳定的基础库。
安装和快速入门
安装owmeta后,您需要连接到一个数据库才能开始使用。OpenWorm owmeta项目当前托管在https://github.com/openworm/OpenWormData.git。您可以通过以下命令克隆该项目:
owm clone https://github.com/openworm/OpenWormData.git --branch owmeta
这将在当前工作目录下创建一个.owm目录。然后,您可以在Python中连接到该项目:
from owmeta_core.command import OWM
conn = OWM().connect()
更多详细的使用说明和示例可以在owmeta-core文档和owmeta Git仓库的examples目录中找到。
贡献和支持
owmeta是一个开源项目,欢迎社区贡献。如果您想参与贡献,可以提交pull请求或报告bug。如果您不确定如何贡献,可以填写这个短表单,您将收到加入我们Slack聊天的邀请,在那里可以进行更深入的交流。
如果您有任何问题、疑虑或功能建议,也可以在项目的issue tracker上提出。
结语
owmeta为C. elegans研究提供了一个强大而灵活的数据访问工具。通过整合多种数据表示和提供统一的接口,它大大简化了研究人员获取和分析复杂生物学数据的过程。随着OpenWorm项目的不断发展,owmeta将继续发挥关键作用,推动我们对这种模式生物的理解不断深入。🧬🔬









