UGENE: 开源跨平台生物信息学软件工具包
UGENE简介
UGENE是一款免费开源的跨平台生物信息学软件工具包,由Unipro公司开发并维护。作为一个集成化的平台,UGENE为分子生物学家和生物信息学研究人员提供了丰富的分析工具和直观的图形用户界面,极大地简化了复杂生物数据的处理和分析流程。
自2008年首次发布以来,UGENE经过十多年的持续开发和完善,目前已成为生物信息学领域广受欢迎的软件之一。它支持Windows、macOS和Linux等主流操作系统,为用户提供了良好的跨平台兼容性。

主要功能特性
UGENE集成了大量常用的生物信息学工具和算法,主要功能包括但不限于:
- DNA/RNA和蛋白质序列的创建、编辑和注释
- 多序列比对(支持ClustalW/O、MUSCLE、MAFFT等算法)
- 系统发育树构建和编辑
- 基因组浏览和注释
- 引物设计
- 限制性内切酶位点分析
- 序列搜索和比对(如BLAST)
- 二代测序数据分析(包括reads比对、变异检测等)
- 3D蛋白质结构可视化
此外,UGENE还提供了工作流设计器,允许用户构建可重复使用的分析流程,大大提高了复杂实验的效率。
用户友好的界面设计
作为面向生物学家的软件,UGENE在用户界面设计上做了大量优化,以确保即使是编程经验有限的研究人员也能轻松上手。其主要特点包括:
- 图形化操作界面,降低了学习门槛
- 项目管理功能,方便组织和管理分析数据
- 丰富的数据可视化组件,包括序列查看器、多序列比对编辑器、系统发育树查看器等
- 直观的工作流设计器,支持拖拽式操作
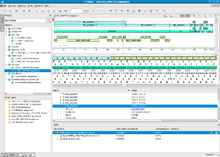
高性能计算支持
为了提高大规模数据分析的效率,UGENE对部分算法进行了优化,以充分利用现代计算机硬件的性能:
- 多线程并行计算,提高CPU利用率
- 支持GPU加速计算(如Smith-Waterman算法)
- 针对特定处理器指令集进行优化
这些优化使得UGENE能够高效处理大型基因组数据和海量测序数据。
扩展性和可定制性
UGENE采用模块化设计,具有良好的扩展性:
- 插件系统允许开发者添加新功能
- 工作流设计器支持自定义分析流程
- 命令行界面便于集成到其他分析管道中
- 支持通过脚本语言(如Python)扩展功能
这种灵活的架构使得UGENE能够适应不同研究团队的需求。
社区支持和持续开发
作为一个开源项目,UGENE拥有活跃的用户社区和开发团队:
- GitHub上的源代码仓库开放贡献
- 定期发布新版本,持续改进功能和性能
- 详细的在线文档和教程视频
- 用户论坛提供技术支持和交流
社区的参与确保了UGENE能够紧跟生物信息学领域的最新发展。
应用领域
UGENE在多个生物学研究领域得到广泛应用,包括:
- 基因组学和转录组学研究
- 进化生物学和系统发育分析
- 蛋白质结构预测和分析
- 分子生物学实验设计(如PCR引物设计)
- 病毒学和微生物组学研究
总结
UGENE作为一款功能全面、用户友好的生物信息学软件工具包,为生命科学研究提供了强大的数据分析支持。它不仅集成了大量常用工具和算法,还通过图形化界面和工作流系统大大简化了复杂分析流程的设计和执行。作为一个持续发展的开源项目,UGENE有望在未来继续为生物信息学领域做出重要贡献。
研究人员可以通��过访问UGENE官方网站了解更多信息,下载软件并参与到这个开源项目的开发中来。随着生物数据规模的不断扩大和分析需求的日益复杂,像UGENE这样的集成工具包必将在推动生命科学研究方面发挥越来越重要的作用。
编辑推荐精选


讯飞智文
一键生成PPT和Word,让学习生活更轻松
讯飞智文是一个利用 AI 技术的项目,能够帮助用户生成 PPT 以及各类文档。无论是商业领域的市场分析报告、年度目标制定,还是学生群体的职业生涯规划、实习避坑指南,亦或是活动策划、旅游攻略等内容,它都能提供支持,帮助用户精准表达,轻松呈现各种信息。


讯飞星火
深度推理能力全新升级,全面对标OpenAI o1
科大讯飞的星火大模型,支持语言理解、知识问答和文本创作等多功能,适用于多种文件和业务场景,提升办公和日常生活的效率。讯飞星火是一个提供丰富智能服务的平台,涵盖科技资讯、图像创作、写作辅助、编程解答、科研文献解读等功能,能为不同需求的用户提供便捷高效的帮助,助力用户轻松获取信息、解决问题,满足多样化使用场景。


Spark-TTS
一种基于大语言模型的高效单流解耦语音令牌文本到语音合成模型
Spark-TTS 是一个基于 PyTorch 的开源文本到语音合成项目,由多个知名机构联合参与。该项目提供了高效的 LLM(大语言模型)驱动的语音合成方案,支持语音克隆和语音创建功能,可通过命令行界面(CLI)和 Web UI 两种方式使用。用户可以根据需求调整语音的性别、音高、速度等参数,生成高质量的语音。该项目适用于多种场景,如有声读物制作、智能语音助手开发等。


Trae
字节跳动发布的AI编程神器IDE
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


咔片PPT
AI助力,做PPT更简单!
咔片是一款轻量化在线演示设计工具,借助 AI 技术,实现从内容生成到智能设计的一站式 PPT 制作服务。支持多种文档格式导入生成 PPT,提供海量模板、智能美化、素材替换等功能,适用于销售、教师、学生等各类人群,能高效制作出高品质 PPT,满足不同场景演示需求。


讯飞绘文
选题、配图、成文,一站式创作,让内容运营更高效
讯飞绘文,一个AI集成平台,支持写作、选题、配图、排版和发布。高效生成适用于各类媒体的定制内容,加速品牌传播,提升内容营销效果。


材料星
专业的AI公文写作平台,公文写作神器
AI 材料星,专业的 AI 公文写作辅助平台,为体制内工作人员提供高效的公文写作解决方案。拥有海量公文文库、9 大核心 AI 功能,支持 30 + 文稿类型生成,助力快速完成领导讲话、工作总结、述职报告等材料,提升办公效率,是体制打工人的得力写作神器。


openai-agents-python
OpenAI Agents SDK,助力开发者便捷使用 OpenAI 相关功能。
openai-agents-python 是 OpenAI 推出的一款强大 Python SDK,它为开发者提供了与 OpenAI 模型交互的高效工具,支持工具调用、结果处理、追踪等功能,涵盖多种应用场景,如研究助手、财务研究等,能显著提升开发效率,让开发者更轻松地利用 OpenAI 的技术优势。


Hunyuan3D-2
高分辨率纹理 3D 资产生成
Hunyuan3D-2 是腾讯开发的用于 3D 资产生成的强大工具,支持从文本描述、单张图片或多视角图片生成 3D 模型,具备快速形状生成能力,可生成带纹理的高质量 3D 模型,适用于多个领域,为 3D 创作提供了高效解决方案。


3FS
一个具备存储、管理和客户端操作等多种功能的分布式文件系统相关项目。
3FS 是一个功能强大的分布式文件系统项目,涵盖了存储引擎、元数据管理、客户端工具等多个模块。它支持多种文件操作,如创建文件和目录、设置布局等,同时具备高效的事件循环、节点选择和协程池管理等特性。适用于需要大规模数据存储和管理的场景,能够提高系统的性能和可靠性,是分布式存储领域的优质解决方案。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号




