基于人工智能的蛋白质设计精选
这是一个关于基于人工智能的蛋白质设计的研究论文集。 该仓库将持续更新,以跟踪基于人工智能的蛋白质设计的前沿进展。
欢迎关注和点赞!
目录
-
- Nature (<font color="red">重要!!!</font>)
- Nature Biomedical Engineering
- Nature Communications
- Nature Machine Intelligence
- Science
- ICML, ICLR 或 NeurIPS 2022
- Arxiv 或 bioRxiv
- 其他
蛋白质设计概述
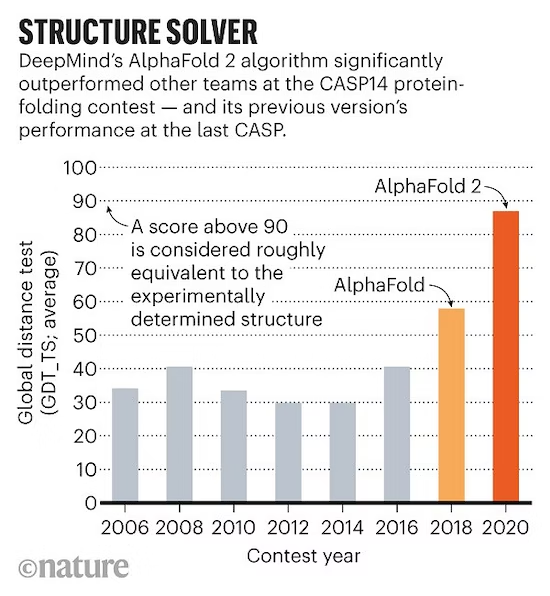
人工智能工具已经解决了蛋白质结构预测问题。这个问题从氨基酸序列推导出空间结构,并达到了原子级别的预测精度,例如AlphaFold 2。它结合了之前的蛋白质结构预测模型,自动学习蛋白质设计方法,从而真正服务于人类的制药需求。
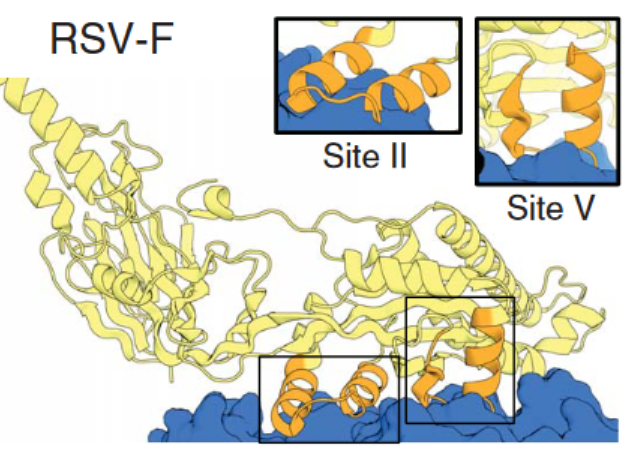
蛋白质设计的具体实践方式多种多样,适用于不同设计过程的问题定义也大不相同。以下是一些例子:
- 从空间结构预测氨基酸序列的问题(Alphafold的逆问题),这假设可以通过分子动力学模拟等方法推导出所需蛋白质的空间结构。
- 对给定部分结构的蛋白质结构完成问题,如著名的David Baker团队最近在Science上发表的论文[1]。这假设只能找到部分结构匹配。
- 将拟合的能量函数与MD模拟相结合进行蛋白质设计,如中国刘海燕团队最近在Nature上发表的论文[2]。
此外,许多方法可用于蛋白质设计,相应的人工智能问题定义也大不相同。本文列出了一些基于人工智能的蛋白质设计的高水平文章,未来将持续更新。
论文
格式:
- [标题](论文链接) [链接]
- 作者1、作者2和作者3...
- 发表方
- 关键词
Nature
-
- Josh Abramson、Jonas Adler、Jack Dunger、Richard Evans、Tim Green、Alexander Pritzel、Olaf Ronneberger、Lindsay Willmore、Andrew J. Ballard、Joshua Bambrick、Sebastian W. Bodenstein、David A. Evans、Chia-Chun Hung、Michael O'Neill、David Reiman、Kathryn Tunyasuvunakool、Zachary Wu、Akvilė Žemgulytė、Eirini Arvaniti、Charles Beattie、Ottavia Bertolli、Alex Bridgland、Alexey Cherepanov、Miles Congreve、Alexander I. Cowen-Rivers、Andrew Cowie、Michael Figurnov、Fabian B. Fuchs、Hannah Gladman、Rishub Jain、Yousuf A. Khan、Caroline M. R. Low、Kuba Perlin、Anna Potapenko、Pascal Savy、Sukhdeep Singh、Adrian Stecula、Ashok Thillaisundaram、Catherine Tong、Sergei Yakneen、Ellen D. Zhong、Michal Zielinski、Augustin Žídek、Victor Bapst、Pushmeet Kohli、Max Jaderberg、Demis Hassabis 和 John M. Jumper
- 关键词:基于扩散的架构、蛋白质结构建模、生物分子空间建模
-
- B Huang、Y Xu、X Hu、Y Liu、S Liao、J Zhang、C Huang
- 关键词:能量函数、MD模拟、以骨架为中心
-
- Ivan Anishchenko、Samuel J. Pellock、Tamuka M. Chidyausiku、Theresa A. Ramelot、Sergey Ovchinnikov、Jingzhou Hao、Khushboo Bafna、Christoffer Norn、Alex Kang、Asim K. Bera、Frank DiMaio、Lauren Carter、Cameron M. Chow、Gaetano T. Montelione 和 David Baker
- 关键词:幻觉、修复、蛋白质设计
-
- Longxing Cao、Brian Coventry、Inna Goreshnik、Buwei Huang、William Sheffler、Joon Sung Park、Kevin M. Jude、Iva Marković、Rameshwar U. Kadam、Koen H. G. Verschueren、Kenneth Verstraete、Scott Thomas Russell Walsh、Nathaniel Bennett、Ashish Phal、Aerin Yang、Lisa Kozodoy、Michelle DeWitt、Lora Picton、Lauren Miller、Eva-Maria Strauch、Nicholas D. DeBouver、Allison Pires、Asim K. Bera、Samer Halabiya、Bradley Hammerson、Wei Yang、Steffen Bernard、Lance Stewart、Ian A. Wilson、Hannele Ruohola-Baker、Joseph Schlessinger、Sangwon Lee、Savvas N. Savvides、K. Christopher Garcia 和 David Baker
- 关键词:结合位点
Nature Biomedical Engineering
- 通过深度生成模型和分子动力学模拟加速抗菌剂的发现
- Payel Das、Tom Sercu、Kahini Wadhawan、Inkit Padhi、Sebastian Gehrmann、Flaviu Cipcigan、Vijil Chenthamarakshan、Hendrik Strobelt、Cicero dos Santos、Pin-Yu Chen、Yi Yan Yang、Jeremy P. K. Tan、James Hedrick、Jason Crain 和 Aleksandra Mojsilovic
- 关键词:抗菌剂、生成自编码器、分子动力学
Nature Communications
-
- Lorillee Tallorin、JiaLei Wang、Woojoo E. Kim、Swagat Sahu、Nicolas M. Kosa、Pu Yang、Matthew Thompson、Michael K. Gilson、Peter I. Frazier、Michael D. Burkart 和 Nathan C. Gianneschi
- 关键词:酶设计、机器学习
-
- Yunan Luo, Guangde Jiang, Tianhao Yu, Yang Liu, Lam Vo, Hantian Ding, Yufeng Su, Wesley Wei Qian, Huimin Zhao & Jian Peng
- 关键词:功能适应性、进化
-
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse & Debora S. Marks
- 关键词:自回归生成模型、蛋白质设计
-
- Namrata Anand, Raphael Eguchi, Irimpan I. Mathews, Carla P. Perez, Alexander Derry, Russ B. Altman & Po-Ssu Huang
- 关键词:蛋白质设计、能量函数、深度神经网络
-
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse & Debora S. Marks
- 关键词:自�回归生成模型、蛋白质设计
Nature Machine Intelligence
-
- Jannis Born & Matteo Manica
- 关键词:分子语言建模、回归、转换器
-
- Noelia Ferruz & Birte Höcker
- 关键词:语言模型、可控、综述
Science
-
- J. Dauparas, I. Anishchenko, N. Bennett, H. Bai, R. J. Ragotte, L. F. Milles, B. I. M. Wicky, A. Courbet, R. J. de Haas, N. Bethel, P. J. Y. Leung, T. F. Huddy, S. Pellock, D. Tischer, F. Chan, B. Koepnick, H. Nguyen, A. Kang, B. Sankaran, A. K. Bera, N. P. King, D. Baker
- 关键词:语言模型、结构预测
-
- Jue Wang, Sidney Lisanza, David Juergens, Doug Tischer, Joseph L. Watson, Karla M. Castro, Robert Ragotte, Amijai Saragovi, Lukas F. Milles, Minkyung Baek, Ivan Anishchenko, Wei Yang, Derrick R. Hicks, Marc Expòsit, Thomas Schlichthaerle, Jung-Ho Chun, Justas Dauparas, Nathaniel Bennett, Basile I. M. Wicky, Andrew Muenks, Frank DiMaio, Bruno Correia, Sergey Ovchinnikov, David Baker
- 关键词:功能位点、深度学习、幻觉、修复
ICML、ICLR或NeurIPS
-
- Ziqi Gao, Tao Feng, Jiaxuan You, Chenyi Zi, Yan Zhou, Chen Zhang, Jia Li
- 关键词:蛋白质复合物结构预测、对接路径预测、策略网络、强化学习
-
BERTology遇上生物学:解释蛋白质语言模型中的注意力机制
- Jesse Vig, Ali Madani, Lav R. Varshney, Caiming Xiong, Richard Socher, Nazneen Fatema Rajani
- 关键词:语言模型、转换器、结构和功能特性
-
- Xiangzhe Kong, Wenbing Huang, Yang Liu
- 关键词:抗体设计、图转换
-
- David Brookes, Hahnbeom Park, Jennifer Listgarten
- 关键词:自适应采样、蛋白质设计
-
- Zeming Lin, Tom Sercu, Yann LeCun
- 关键词:多样性、生成模型、蛋白质设计
-
- Zander Harteveld, Joshua Southern, Michaël Defferrard, Andreas Loukas, Pierre Vandergheynst, Micheal Bronstein, Bruno Correia
- 关键词:变分自编码器、拓扑特征、锐化
-
Fold2Seq:用于蛋白质设计的联合序列(1D)-折叠(3D)嵌入生成模型
- Yue Cao, Payel Das, Vijil Chenthamarakshan, Pin-Yu Chen, Igor Melnyk, Yang Shen
- 关键词:生成模型、蛋白质设计
-
- Namrata Anand, Possu Huang
- 关键词:生成模型、蛋白质设计
-
- John Ingraham, Vikas Garg, Regina Barzilay, Tommi Jaakkola
- 关键词:生成模型、蛋白质设计
-
- XChristof Angermueller, David Dohan, David Belanger, Ramya Deshpande, Kevin Murphy, Lucy Colwell
- 关键词:强化学习、序列设计
-
- Zaixi Zhang, Yaosen Min, Shuxin Zheng, Qi Liu
- 关键词:目标蛋白,结构基序,逐片段生成
-
- Zuobai Zhang, Minghao Xu, Arian Jamasb, Vijil Chenthamarakshan, Aurelie Lozano, Payel Das, Jian Tang
- 关键词:药物发现,药物设计,新分子结构生成模型
Arxiv 或 bioRxiv
-
- Sarah Alamdari, Nitya Thakkar, Rianne van den Berg, Alex Xijie Lu, Nicolo Fusi, Ava Pardis Amini, Kevin K Yang
- 关键词:扩散模型,深度生成模型,蛋白质生成,框架,序列设计
-
- Brian Hie, Salvatore Candido, Zeming Lin, Ori Kabeli, Roshan Rao, Nikita Smetanin, Tom Sercu, Alexander Rives
- 关键词:ESMFold,语言模型,基于能量
-
通过整合结构预测网络和扩散生成模型实现广泛适用且准确的蛋白质设计
- Joseph L. Watson, David Juergens, Nathaniel R. Bennett, Brian L. Trippe, Jason Yim, Helen E. Eisenach, Woody Ahern, Andrew J. Borst, Robert J. Ragotte, Lukas F. Milles, Basile I. M. Wicky, Nikita Hanikel, Samuel J. Pellock, Alexis Courbet, William Sheffler, Jue Wang, Preetham Venkatesh, Isaac Sappington, Susana Vázquez Torres, Anna Lauko, Valentin De Bortoli, Emile Mathieu, Regina Barzilay, Tommi S. Jaakkola, Frank DiMaio, Minkyung Baek, David Baker
- 关键词:扩散,通用深度学习框架,从头设计结合蛋白
-
- Vladimir Gligorijević, Daniel Berenberg, Stephen Ra, Simon Kelow, Kyunghyun Cho
- 关键词:序列降噪自编码器,深度流形采样
-
- Robert Verkuil, Ori Kabeli, Yilun Du, Basile I. M. Wicky, Lukas F. Milles, Justas Dauparas, David Baker, Sergey Ovchinnikov, Tom Sercu, Alexander Rives
- 关键词:ESMFold,语言模型,固定骨架设计
-
- Zeming Lin, Halil Akin, Roshan Rao, Brian Hie, Zhongkai Zhu, Wenting Lu, Allan dos Santos Costa, Maryam Fazel-Zarandi, Tom Sercu, Sal Candido, Alexander Rives
- 关键词:结构预测,语言模型
-
TERMinator:使用三级重复基序的基于结构的蛋白质设计神经网络框架
- Alex J. Li, Vikram Sundar, Gevorg Grigoryan, Amy E. Keating
- ��关键词:蛋白质设计,三级基序
-
- Robert Verkuil, Ori Kabeli, Yilun Du, Basile I. M. Wicky, Lukas F. Milles, Justas Dauparas, David Baker, Sergey Ovchinnikov, Tom Sercu, Alexander Rives
- 关键词:语言模型,蛋白质设计
其他
-
- Alexey Strokach, David Becerra, Carles Corbi-Verge, Albert Perez-Riba, Philip M.Kim
- 关键词:蛋白质设计,图神经网络
-
- Jung-Eun Shin, Adam J. Riesselman, Aaron W. Kollasch, Conor McMahon, Elana Simon, Chris Sander, Aashish Manglik, Andrew C. Kruse & Debora S. Marks
- 关键词:变体预测,蛋白质设计,自回归生成模型
参考文献
[1] Wang, Jue, et al. "使用深度学习搭建蛋白质功能位点"。Science 377.6604 (2022): 387-394.
[2] Huang, Bin, et al. "用于蛋白质设计的以骨架为中心的神经网络能量函数"。Nature 602.7897 (2022): 523-528.
贡献
我们的目标是使这个仓库变得更好。如果您有兴趣贡献,请参考这里的贡献指南。
许可证
基于人工智能的蛋白质设计精选集在 Apache 2.0 许可下发布。
编辑推荐精选


讯飞智文
一键生成PPT和Word,让学习生活更轻松
讯飞智文是一个利用 AI 技术的项目,能够帮助用户生成 PPT 以及各类文档。无论是商业领域的市场分析报告、年度目标制定,还是学生群体的职业生涯规划、实习避坑指南,亦或是活动策划、旅游攻略等内容,它都能提供支持,帮助用户精准表达,轻松呈现各种信息。


讯飞星火
深度推理能力全新升级,全面对标OpenAI o1
科大讯飞的星火大模型,支持语言理解、知识问答和文本创作等多功能,适用于多种文件和业务场景,提升办公和日常生活的效率。讯飞星火是一个提供丰富智能服务的平台,涵盖科技资讯、图像创作、写作辅助、��编程解答、科研文献解读等功能,能为不同需求的用户提供便捷高效的帮助,助力用户轻松获取信息、解决问题,满足多样化使用场景。


Spark-TTS
一种基于大语言模型的高效单流解耦语音令牌文本到语音合成模型
Spark-TTS 是一个基于 PyTorch 的开源文本到语音合成项目,由多个知名机构联合参与。该项目提供了高效的 LLM(大语言模型)驱动的语音合成方案,支持语音克隆和语音创建功能,可通过命令行界面(CLI)和 Web UI 两种方式使用。用户可以根据需求调整语音的性别、音高、速度等参数,生成高质量的语音。该项目适用于多种场景,如有声读物制作、智能语音助手开发等。


Trae
字节跳动发布的AI编程神器IDE
Trae是一种自适应的集成�开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


咔片PPT
AI�助力,做PPT更简单!
咔片是一款轻量化在线演示设计工具,借助 AI 技术,实现从内容生成到智能设计的一站式 PPT 制作服务。支持多种文档格式导入生成 PPT,提供海量模板、智能美化、素材替换等功能,适用于销售、教师、学生等各类人群,能高效制作出高品质 PPT,满足不同场景演示需求。


讯飞绘文
选题、配图、成文,一站式创作,让内容运营更高效
讯飞绘文,一个AI集成平台,支持写作、选题、配图、排版和发布。高效生成适用于各类媒体的定制内容,加速品牌传播,提升内容营销效果。


材料星
专业的AI公文写作平台,公文写作神器
AI 材料星,专业的 AI 公文写作辅助平台,为体制内工作人员提供高效的公文写作解决方案。拥有海量公文文�库、9 大核心 AI 功能,支持 30 + 文稿类型生成,助力快速完成领导讲话、工作总结、述职报告等材料,提升办公效率,是体制打工人的得力写作神器。


openai-agents-python
OpenAI Agents SDK,助力开发者便捷使用 OpenAI 相关功能。
openai-agents-python 是 OpenAI 推出的一款强大 Python SDK,它为开发者提供了与 OpenAI 模型交互的高效工具,支持工具调用、结果处理、追踪等功能,涵盖多种应用场景,如研究助手、财务研究等,能显著提升开发效率,让开发者更轻松地利用 OpenAI 的技术优势。


Hunyuan3D-2
高分辨率纹理 3D 资产生成
Hunyuan3D-2 是腾讯开发的用于 3D 资产生成的强大工具,支持从文本描述、单张图片或多视角图片生成 3D 模型,具备快速形状生成能力,可生成带纹理的高质量 3D 模型,适用于多个领域,为 3D 创作提供了高效解决方案。


3FS
一个具备存储、管理和客户端操作等多种功能的分布式文件系统相关项目。
3FS 是一个功能强大的分布式文件系统项目,涵盖了存储引擎、元数据管理、客户端工具等多个模块。它支持多种文件操作,如创建文件和目录、设置布局等,同时具备高效的事件循环、节点选择和协程池管理等特性。适用于需要大规模数据存储和管理的场景,能够提高系统的性能和可靠性,是分布式存储领域的优质解决方案。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号





