DNABERT-2:开启基因组分析新时代
随着高通量测序技术的快速发展,基因组数据呈爆炸式增长。如何从海量的DNA序列中高效提取有价值的生物学知识,成为当前生物信息学领域的一大挑战。近日,来自西北大学的研究团队开发出DNABERT-2,这是一个针对多物种基因组的高效基础模型,在多项基因组分析任务中展现出卓越的性能。
模型创新:提升效率与效果
DNABERT-2在DNABERT的基础上进行了多项技术创新:
-
将k-mer分词替换为字节对编码(BPE),提高了模型的灵活性。
-
采用Attention with Linear Bias (ALiBi)代替传统的位置编码,更好地处理长序列。
-
引入其他优化技术,如Flash Attention和LoRA,进一步提升模型效率。
这些创新使DNABERT-2在保持高性能的同时,大幅降低了计算资源需求。
GUE:全面的基因组理解评估基准
为了全面评估DNABERT-2的性能,研究团队构建了Genome Understanding Evaluation (GUE)基准。GUE包含28个数据集,涵盖7类任务和4个物种,是目前最全面的基因组理解评估基准之一。
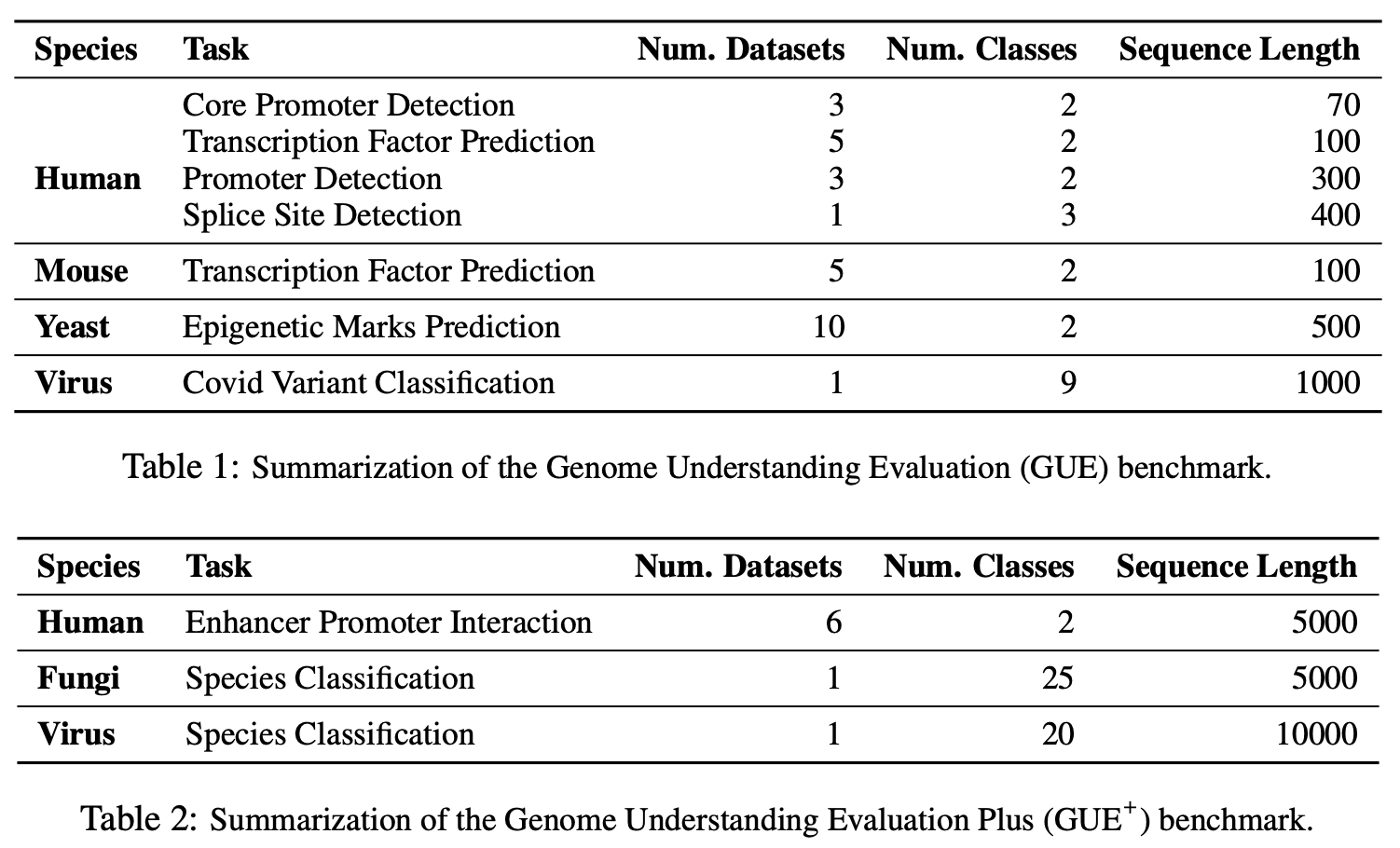
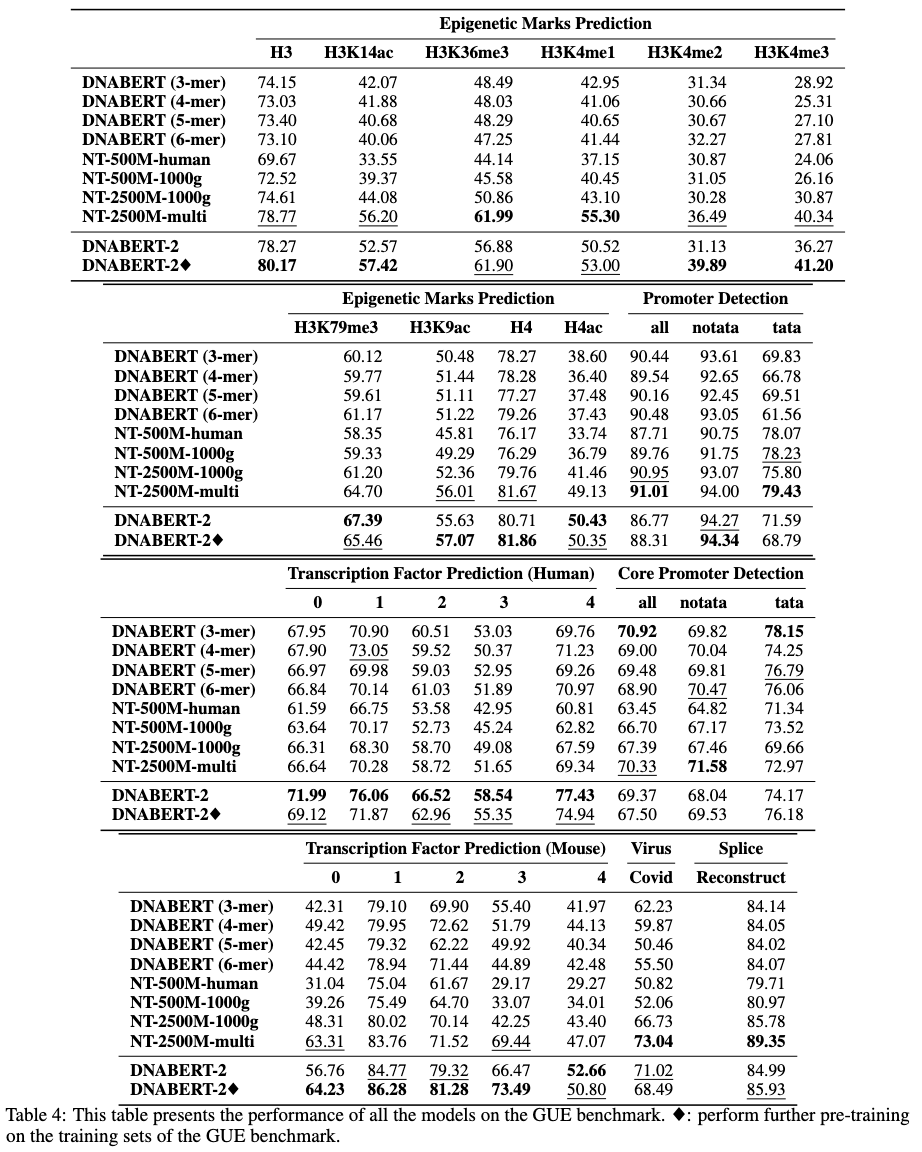
在GUE基准上的评估结果表明,DNABERT-2在多个任务上显著超越了现有方法:
易用性:简化基因组分析流程
DNABERT-2基于广泛使用的Transformers库开发,研究人员只需几行代码即可使用:
from transformers import AutoTokenizer, AutoModel
tokenizer = AutoTokenizer.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True)
model = AutoModel.from_pretrained("zhihan1996/DNABERT-2-117M", trust_remote_code=True)
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0]
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # 768维向量
这种简洁的接口大大降低了使用门槛,使得生物学家也能轻松应用深度学习方法分析基因组数据。
应用前景:赋能多领域基因组研究
DNABERT-2为多个基因组研究领域带来新的可能:
-
基因调控:精准预测转录因子结合位点、启动子区域等调控元件。
-
表观遗传学:分析组蛋白修饰、DNA甲基化等表观遗传标记。
-
基因组变异:识别与疾病相关的DNA变异。
-
进化分析:研究物种间的基因组差异与进化关系。
-
合成生物学:辅助设计人工DNA序列。
此外,DNABERT-2还可以作为其他专门任务模型的预训练基础,进一步提升下游任务的性能。
未来展望:迈向更强大的基因组AI
尽管DNABERT-2取得了显著进展,但基因组AI仍有巨大的发展空间:
-
扩大训练数据:纳入更多物种的基因组数据,提升模型的通用性。
-
优化模型架构:探索更适合DNA序列特点的网络结构。
-
多模态融合:结合蛋白质结构、表达谱等多源信息,实现更全面的基因组理解。
-
可解释性研究:深入分析模型的决策机制,提供生物学洞见。
-
伦理考量:在推进基因组AI的同时,需要充分考虑隐私保护等伦理问题。
DNABERT-2的成功标志着基因组AI进入了一个新的阶段。未来,随着模型的不断优化与数据的持续积累,AI将在揭示生命奥秘、推动精准医疗等方面发挥越来越重要的作用。
结语
DNABERT-2为基因组分析领域带来了新的机遇与挑战。它不仅是一个强大的研究工具,更代表了生物信息学与人工智能深度融合的发展方向。我们期待看到更多研究者基于DNABERT-2开展创新性的工作,共同推动基因组学研究的快速发展。










