Kaggle PANDA竞赛简介
PANDA(Prostate cANcer graDe Assessment)是Kaggle平台上的一项重要医学影像竞赛,旨在利用人工智能技术对前列腺癌组织病理切片进行自动分级。该竞赛吸引了来自全球的3,000多支队伍参与,对推动前列腺癌诊断技术的发展具有重要意义。
本文将详细介绍该竞赛的冠军解决方案,这一方案由Team PND的@yukkyo和@kentaroy47共同开发。他们的模型和代码以CC-BY-NC 4.0许可开源,为整个医学AI社区提供了宝贵的参考。
环境配置
冠军团队提供了两种环境配置方式:
-
不使用Docker(未经测试):
- Ubuntu 18.04
- Python 3.7.2
- CUDA 10.2
- NVIDIA/apex == 1.0
-
使用Docker(推荐):
# 构建 $ sh docker/build.sh # 运行 $ sh docker/run.sh # 执行 $ sh docker/exec.sh
使用Docker可以确保环境的一致性,是复现结果的首选方式。
数据准备
数据准备是整个解决方案的基础,主要包括以下步骤:
-
获取数据:仅下载train_images和train_masks。
-
图像哈希分组:使用图像哈希技术检测重复图像并进行分组。输出文件为
input/duplicate_imgids_imghash_thres_090.csv。 -
K折交叉验证划分:
$ cd src $ python data_process/s00_make_k_fold.py输出文件为
input/train-5kfold.csv。 -
制作训练用的图像块:
$ cd src $ python data_process/s07_simple_tile.py --mode 0 $ python data_process/s07_simple_tile.py --mode 2 $ python data_process/a00_save_tiles.py
这些步骤为后续的模型训练奠定了坚实的数据基础。
基础模型训练
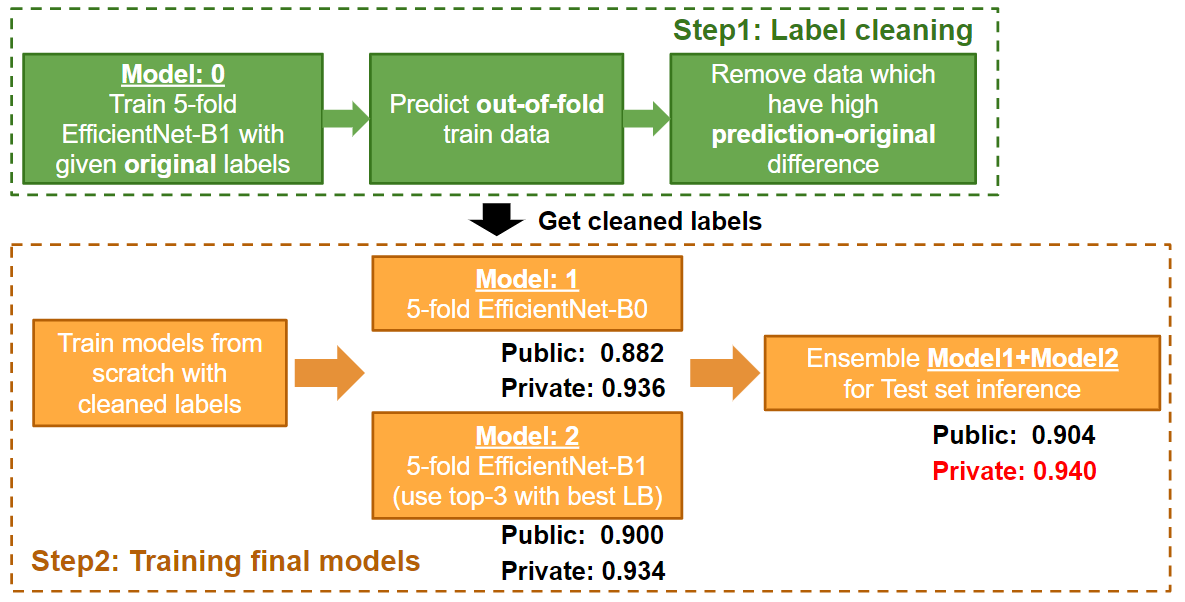
基础模型的训练是为了去除数据中的噪声,这一步骤至关重要:
$ cd src
$ python train.py --config configs/final_1.yaml --kfold 1
$ python train.py --config configs/final_1.yaml --kfold 2
$ python train.py --config configs/final_1.yaml --kfold 3
$ python train.py --config configs/final_1.yaml --kfold 4
$ python train.py --config configs/final_1.yaml --kfold 5
每个fold大约需要18小时,输出将保存在output/model/final_1目录下。
本地验证预测
训练完成后,需要对本地验证集进行预测,以便后续的去噪处理:
$ cd src
$ python kernel.py --kfold 1
$ python kernel.py --kfold 2
$ python kernel.py --kfold 3
$ python kernel.py --kfold 4
$ python kernel.py --kfold 5
每个fold大约需要1小时,预测结果将保存为output/model/final_1/local_preds~~~.csv。
去除噪声
去噪是提高模型性能的关键步骤:
$ cd src
$ python data_process/s12_remove_noise_by_local_preds.py
这一步骤会生成多个重要的CSV文件:
local_preds_final_1_efficientnet-b1.csv: 用于清理标签的预测结果。local_preds_final_1_efficientnet-b1_removed_noise_thresh_16.csv: 用于训练Model 1的基础标签清理结果。local_preds_final_1_efficientnet-b1_removed_noise_thresh_rad_13_08_ka_15_10.csv: 用于训练Model 2的标签,移除了20%的Radboud标签。
重新训练去噪后的模型
在去除噪声后,需要重新训练模型以获得更好的性能:
-
训练Model 2(fam_taro模型):
$ cd src $ python train.py --config configs/final_2.yaml --kfold 1 $ python train.py --config configs/final_2.yaml --kfold 4 $ python train.py --config configs/final_2.yaml --kfold 5 -
训练Model 1(arutema模型): 运行
train_famdata-kfolds.ipynb或train_famdata-kfolds.py。
每个fold的训练时间约为4-15小时不等。最终的模型将保存在models目录中。
Kaggle提交
最后一步是在Kaggle Notebook上提交结果:
-
冠军方案最终提交:
- 公共得分: 0.904
- 私有得分: 0.940 (第1名)
- 提交链接
-
可复现的结果(固定随机种子):
- 公共得分: 0.894
- 私有得分: 0.939 (第1名)
- 提交链接
-
简单的5折模型(第3名):
- 私有得分: 0.935
- 提交链接
总结与展望
Team PND的解决方案不仅在Kaggle竞赛中取得了优异成绩,还被多个知名学术期刊采用,包括:
- Nature Medicine: W.Bulten等人的研究
- npj Precision Oncology: Y.Tolkach等人的验证研究
- Cancers: 基于标签分布学习的自动癌症分级研究
这些成果充分证明了该解决方案在实际医学应用中的潜力。未来,我们可以期待这一技术在以下方面的进一步发展:
-
模型轻量化:优化模型结构,减少计算资源需求,使其更适合临床实时应用。
-
多模态融合:结合其他类型的医学影像数据(如MRI、CT等),提高诊断的准确性和全面性。
-
可解释性研究:深入探究模型的决策过程,提高医生对AI辅助诊断的信任度。
-
泛化能力提升:在更多样化的数据集上进行测试和优化,确保模型在不同医疗机构和人群中的适用性。
-
临床试验验证:与医疗机构合作,开展大规模临床试验,进一步验证模型在实际诊断中的效果。
总的来说,Kaggle PANDA竞赛的这个冠军解决方案不仅展示了AI在医学影像分析中的巨大潜力,也为未来的研究指明了方向。随着技术的不断进步和跨学科合作的深入,我们有理由相信,AI辅助诊断将在前列腺癌以及其他疾病的早期发现和精准治疗中发挥越来越重要的作用。









