OpenFold:开源实现引领蛋白质结构预测新时代
在生物学研究中,蛋白质结构预测一直是一个具有挑战性的问题。2020年,DeepMind公司的AlphaFold 2在这一领域取得了突破性进展,将预测精度提升到了接近实验方法的水平。然而,AlphaFold 2的代码和训练数据并未完全开放,这在一定程度上限制了科研界对其进行深入研究和改进。为了推动这一技术的进一步发展,来自哥伦比亚大学、哈佛医学院等机构的研究人员开发了OpenFold项目,这是一个完全开源、可训练的AlphaFold 2复现版本。
OpenFold的主要特点
OpenFold不仅仅是对AlphaFold 2的简单复制,它在多个方面都有创新和改进:
-
完全开源: OpenFold的全部代码和训练数据都是公开的,这为研究人员提供了深入研究和改进算法的机会。
-
可训练性: 与原版AlphaFold 2不同,OpenFold提供了完整的训练管道,允许研究者使用自己的数据集重新训练模型。
-
高效推理: 在GPU上,OpenFold的推理速度可以比原版快2倍,特别是在Ampere或更高架构的GPU上表现突出。
-
内存优化: 通过实现低内存注意力机制和自定义CUDA注意力核心,OpenFold大大降低了GPU内存需求,使其能够处理超长序列(超过4000个残基)的蛋白质。
-
多种精度支持: OpenFold支持全精度、半精度或bfloat16精度训练,并可选择是否使用DeepSpeed进行加速。
-
兼容性: OpenFold支持使用AlphaFold的官方参数进行推理,反之亦然,保证了与现有研究的兼容性。
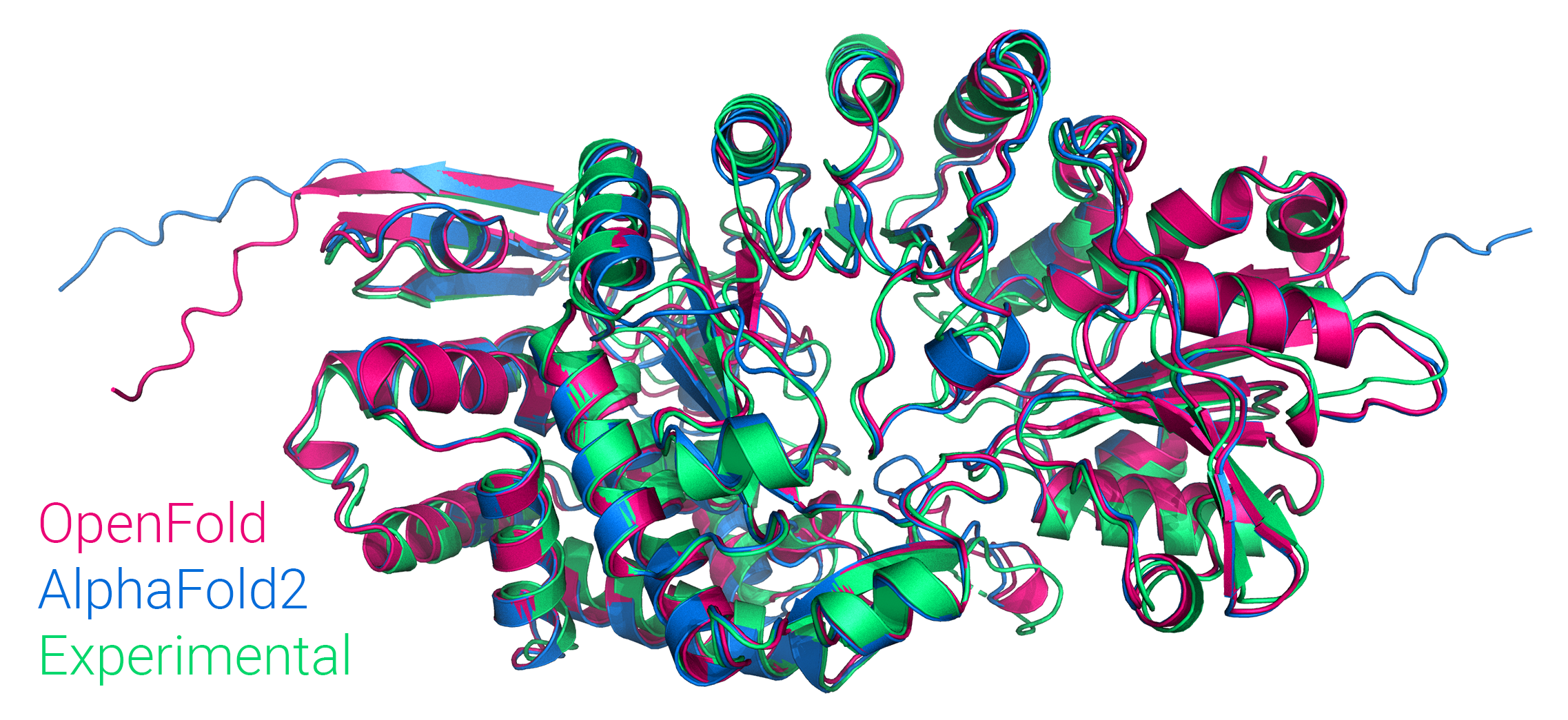 图1: OpenFold与AlphaFold2对PDB 7KDX链B的预测结果与实验结构的比较
图1: OpenFold与AlphaFold2对PDB 7KDX链B的预测结果与实验结构的比较
OpenFold的技术创新
OpenFold在实现过程中融入了多项技术创新,进一步提升了模型的性能和灵活性:
-
低内存注意力机制: 基于Rabe & Staats 2021年的研究,OpenFold实现了低内存注意力机制,使其能够处理超长序列。
-
自定义CUDA注意力核心: 通过修改FastFold的核心,OpenFold实现了推理和训练过程中的原位注意力计算,比FastFold和标准PyTorch实现分别节省了4倍和5倍的GPU内存。
-
高效对齐脚本: OpenFold提供了使用原始AlphaFold HHblits/JackHMMER管道或更快的MMseqs2(来自ColabFold)的高效对齐脚本。
-
FlashAttention支持: 通过集成FlashAttention,大大加速了MSA(多序列比对)注意力计算。
-
DeepSpeed DS4Sci_EvoformerAttention核心: 作为与DeepSpeed4Science倡议合作的成果,这一内存高效的注意力核心显著提升了训练和推理速度,同时将模型的峰值设备内存需求降低了13倍。
OpenFold的应用前景
OpenFold的出现为蛋白质结构预测领域带来了新的可能性:
-
学术研究: 研究人员可以深入了解模型的工作原理,探索改进空间,推动算法的进一步发展。
-
药物研发: 制药公司可以利用OpenFold预测新药物靶点的结构,加速药物发现过程。
-
个性化医疗: 通过预测患者特定蛋白质的结构,有助于开发更精准的治疗方案。
-
生物工程: 在蛋白质设计和优化中,OpenFold可以提供valuable insights,辅助创造具有特定功能的新型蛋白质。
-
教育培训: OpenFold为学生和研究人员提供了一个学习和实践最先进蛋白质结构预测技术的平台。
未来展望
虽然OpenFold已经取得了令人瞩目的成果,但蛋白质结构预测领域仍有很大的发展空间。未来的研究方向可能包括:
-
进一步提高预测精度: 特别是对于一些难以预测的蛋白质家族。
-
扩展到更复杂的生物大分子系统: 如蛋白质-蛋白质复合物、蛋白质-核酸复合物等。
-
整合实验数据: 结合低分辨率实验数据来提高预测的准确性。
-
动态结构预测: 不仅预测静态结构,还能模拟蛋白质的动态行为。
-
与其他AI技术的结合: 如将大型语言模型与结构预测模型相结合,提供更全面的蛋白质功能预测。
OpenFold的开源性质意味着这些进展可能来自全球各地的研究者,形成一个协作共赢的生态系统。这种开放式创新模式有望加速蛋白质科学的发展,为生物学、医学和生物技术带来更多突破性进展。
总的来说,OpenFold代表了蛋白质结构预测领域的一个重要里程碑。它不仅复现了AlphaFold 2的卓越性能,还通过开源和技术创新为整个领域注入了新的活力。随着越来越多的研究者加入这个开放的平台,我们有理由期待在不久的将来,蛋白质结构预测技术将迎来更多令人兴奋的突破。









