SLANTbrainSeg简介
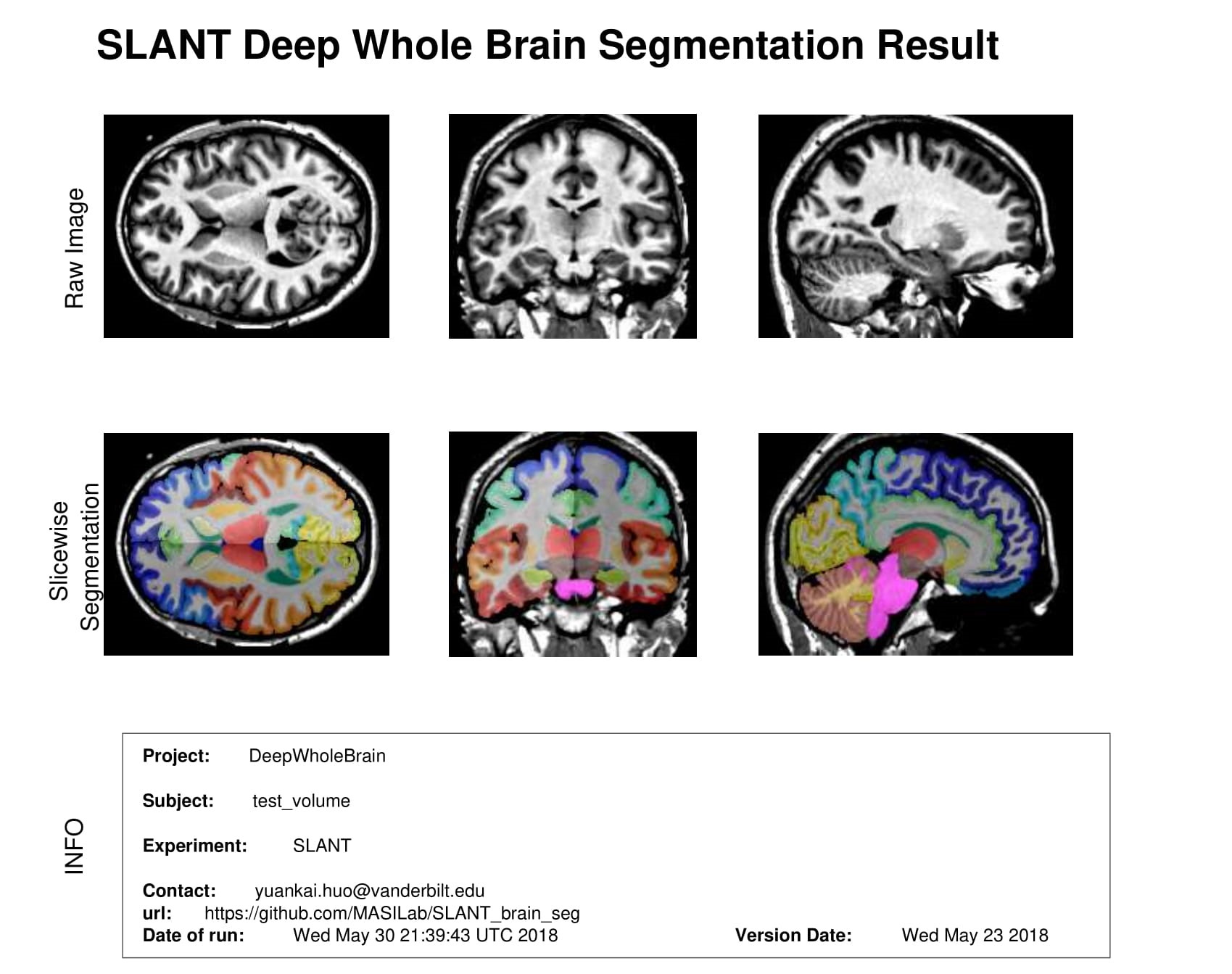
SLANTbrainSeg(Spatially Localized Atlas Network Tiles)是由Vanderbilt大学医学影像科学研究所(MASI)开发的一种全新的全脑分割算法。该方法可以将T1 MRI扫描精确分割为133个解剖标签,基于BrainCOLOR协议。SLANTbrainSeg结合了空间定位和深度学习技术,实现了高效准确的全脑组织分割。
主要特点
SLANTbrainSeg具有以下几个主要特点:
-
高精度分割:可将大脑分割为133个解剖标签,精度高。
-
基于深度学习:采用先进的深度学习算法,提高了分割的准确性和效率。
-
空间定位技术:结合空间信息,提高了分割的局部一致性。
-
Docker封装:提供了封装好的Docker镜像,便于部署和使用。
-
支持GPU/CPU:既可以使用GPU加速,也支持纯CPU环境运行。
-
开源可用:代码开源,支持二次开发。
技术原理
SLANTbrainSeg的核心思想是将整个大脑分割任务分解为多个局部分割子任务。具体来说,它包含以下几个关键步骤:
-
预处理:对输入的T1 MRI图像进行预处理,如去除头骨、强度归一化等。
-
空间定位:将大脑空间划分为多个重叠的局部区域(tiles)。
-
深度学习分割:对每个局部区域应用3D卷积神经网络进行分割。
-
后处理:将各个局部区域的分割结果进行融合,得到最终的全脑分割。
这种方法既保证了全局一致性,又提高了局部细节的分割精度。
使用方法
SLANTbrainSeg提供了简单易用的Docker镜像,使用步骤如下:
- 拉取Docker镜像:
sudo docker pull masidocker/public:deep_brain_seg_v1_1_0
- 准备输入数据:
export input_dir=/path/to/input
sudo mkdir $input_dir
sudo wget -O $input_dir/test_volume.nii.gz https://www.nitrc.org/frs/download.php/10666/test_volume.nii.gz
- 运行分割:
export output_dir=$input_dir/output
sudo nvidia-docker run -it --rm -v $input_dir:/INPUTS/ -v $output_dir:/OUTPUTS masidocker/public:deep_brain_seg_v1_1_0 /extra/run_deep_brain_seg.sh
- 查看结果:
- 分割结果位于"FinalResult"文件夹
- 叠加PDF位于"FinalPDF"文件夹
- 体积统计结果位于"FinalVolTxt"文件夹
SLANTbrainSeg还提供了CPU版本的Docker镜像,适用于没有GPU的环境:
sudo docker pull masidocker/public:deep_brain_seg_v1_1_0_CPU
性能评估
研究人员对SLANTbrainSeg进行了全面的性能评估。在OASIS数据集上的实验结果表明:
- SLANTbrainSeg的分割精度优于传统的多图谱配准方法
- 相比单一的3D U-Net,SLANTbrainSeg显著提高了分割的局部一致性
- 在NVIDIA Titan GPU上,分割单个全脑体积仅需约15分钟
详细的评估结果可在NeuroImage论文中查看。
应用前景
SLANTbrainSeg为脑影像研究和临床应用提供了强大的工具,具有广阔的应用前景:
-
神经科学研究:精确量化大脑不同区域的体积和形态。
-
神经系统疾病诊断:辅助诊断阿尔茨海默症、帕金森病等神经退行性疾病。
-
脑外科手术规划:为脑肿瘤切除等手术提供精确的解剖信息。
-
纵向研究:追踪大脑随时间的变化,研究大脑发育和衰老过程。
-
群体分析:进行大规模的脑影像队列研究,揭示疾病相关的脑结构改变。
总结与展望
SLANTbrainSeg作为一种创新的全脑分割方法,结合了空间定位和深度学习技术,实现了高效准确的全脑组织分割。它不仅在分割精度上超越了传统方法,而且提供了便捷的使用方式,为脑影像研究和临床应用带来了新的可能。
未来,SLANTbrainSeg团队将继续优化算法,提高分割速度和精度。同时,他们也欢迎社区贡献,共同推动这一开源项目的发展。随着深度学习技术的进步,我们可以期待SLANTbrainSeg在未来为脑科学研究和临床实践带来更多突破性的进展。
🧠💻 如果您对SLANTbrainSeg感兴趣,可以访问项目GitHub页面获取更多信息,并尝试使用这一强大的脑分割工具!









