ChromBPNet简介
ChromBPNet是由斯坦福大学Kundaje实验室开发的一种革新性深度学习模型,用于分析和预测染色质可及性。染色质可及性是理解基因调控的关键,而ChromBPNet通过其独特的设计,为这一领域带来了新的洞察。
背景与意义
染色质可及性反映了DNA在细胞核中的打开状态,是转录因子结合和基因表达调控的重要指标。传统的实验方法如ATAC-seq和DNase-seq可以测量染色质可及性,但这些方法存在一些局限性,如酶偏差和分辨率不足等问题。ChromBPNet的出现正是为了解决这些挑战。
ChromBPNet的创新设计
ChromBPNet采用了一种全卷积神经网络架构,具有以下几个主要创新点:
-
偏差因子分解:ChromBPNet通过一个两步过程自动校正实验偏差。首先学习一个简单的背景模型来捕捉酶的影响,然后利用这个模型从ATAC-seq/DNase-seq数据中去除酶的效应。
-
碱基分辨率预测:利用膨胀卷积和残差连接,ChromBPNet能够在保持高效参数化的同时实现大感受野,从而提供碱基级别的高分辨率预测。
-
多分辨率建模:ChromBPNet可以捕捉染色质可及性profile的多尺度特征,反映转录因子协同结合的复杂调控模式。
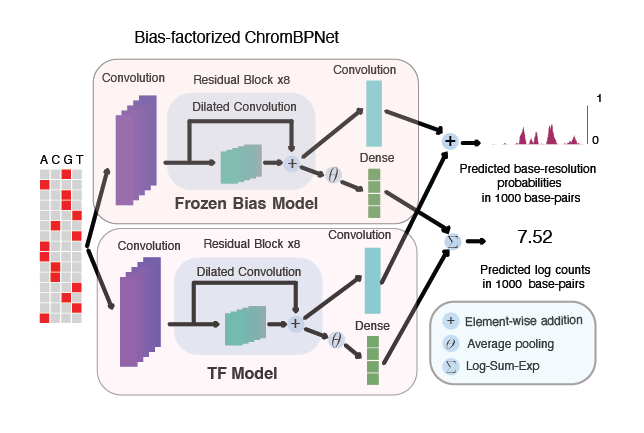
上图展示了ChromBPNet的整体架构,包括偏差模型(Frozen Bias Model)和序列模型(TF Model)两个主要组成部分。
ChromBPNet的应用与优势
ChromBPNet为研究人员提供了强大的工具,可以应用于多个方面:
-
顺式调控序列语法解析:ChromBPNet可以识别和解释影响染色质可及性的DNA序列模式。
-
转录因子足迹分析:通过高分辨率预测,ChromBPNet能够揭示转录因子在染色质上的结合位点和模式。
-
调控变异体鉴定:ChromBPNet可以预测DNA序列变异对染色质可及性的影响,有助于发现功能性的调控变异。
-
跨物种比较:由于ChromBPNet是基于序列的模型,它可以应用于不同物种的比较分析,揭示保守和特异的调控机制。
ChromBPNet的这些应用为表观遗传学和基因调控研究提供了新的视角和方法。
ChromBPNet的使用
安装
ChromBPNet可以通过Docker或本地安装两种方式使用。对于大多数用户,推荐使用Docker方式,因为它可以确保所有依赖项都正确安装:
docker run -it --rm --memory=100g --gpus device=0 kundajelab/chrombpnet:latest
对于希望本地安装的用户,可以按照以下步骤操作:
- 创建conda环境:
conda create -n chrombpnet python=3.8
conda activate chrombpnet
- 安装非Python依赖:
conda install -y -c conda-forge -c bioconda samtools bedtools ucsc-bedgraphtobigwig pybigwig meme
- 从源代码安装ChromBPNet:
git clone https://github.com/kundajelab/chrombpnet.git
pip install -e chrombpnet
模型训练
ChromBPNet的训练分为两个主要步骤:偏差模型训练和完整ChromBPNet模型训练。
- 偏差模型训练:
chrombpnet bias pipeline \
-ibam /path/to/input.bam \
-d "ATAC" \
-g /path/to/hg38.fa \
-c /path/to/hg38.chrom.sizes \
-p /path/to/peaks.bed \
-n /path/to/nonpeaks.bed \
-fl /path/to/fold_0.json \
-b 0.5 \
-o path/to/output/dir/
- 完整ChromBPNet模型训练:
chrombpnet pipeline \
-ibam /path/to/input.bam \
-d "ATAC" \
-g /path/to/hg38.fa \
-c /path/to/hg38.chrom.sizes \
-p /path/to/peaks.bed \
-n /path/to/nonpeaks.bed \
-fl /path/to/fold_0.json \
-b /path/to/bias.h5 \
-o path/to/output/dir/
这些命令中的参数需要根据实际数据和需求进行调整。详细的参数说明和最佳实践可以在ChromBPNet的文档中找到。
ChromBPNet的输出与解释
ChromBPNet的输出包含多个文件,提供了丰富的信息:
- 模型文件:包括偏差模型和完整的ChromBPNet模型。
- 日志文件:记录了训练过程中的损失值和其他参数。
- 评估报告:包括模型性能的各种指标和可视化结果。
- 预测结果:包括碱基分辨率的可及性预测和转录因子结合位点预测。
研究人员可以利用这些输出进行深入的分析,如识别重要的调控元件、预测转录因子结合位点、分析DNA序列变异的影响等。
结论与展望
ChromBPNet代表了染色质可及性分析领域的一个重要进展。通过结合深度学习和生物学知识,它为研究人员提供了一个强大的工具,可以更精确地理解基因调控的复杂性。随着更多数据的积累和算法的进一步优化,我们可以期待ChromBPNet在未来能够:
- 整合更多类型的组学数据,如转录组、表观基因组等。
- 应用于单细胞水平的分析,揭示细胞异质性中的调控差异。
- 扩展到更多物种,促进比较基因组学研究。
- 与其他机器学习方法结合,如强化学习或图神经网络,以捕捉更复杂的调控模式。
ChromBPNet的发展无疑将推动基因调控研究的深入,为精准医疗和基因治疗等领域提供重要的理论基础。研究人员应当密切关注这一工具的更新和应用,以便在自己的研究中充分利用其潜力。









