SynthSeg
在本仓库中,我们介绍SynthSeg,这是第一个可以对任何对比度和分辨率的脑部扫描进行分割的深度学习工具。SynthSeg无需任何再训练即可直接使用,并且对以下因素具有鲁棒性:
- 任何对比度
- 任何分辨率,最高可达10mm切片间距
- 广泛的人群:从年轻健康到老年和患病人群
- 有预处理或无预处理的扫描:偏场校正、颅骨剥离、标准化等
- 白质病变
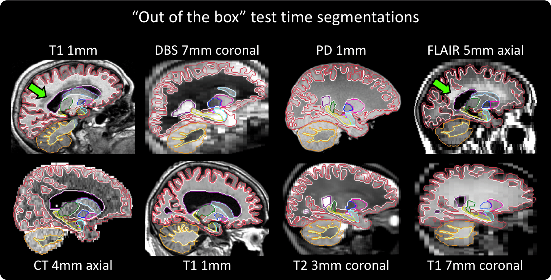
SynthSeg最初是为自动分割任何对比度和分辨率的脑部扫描而设计的。
SynthSeg:无需再训练即可分割任何对比度和分辨率的大脑MRI扫描
B. Billot, D.N. Greve, O. Puonti, A. Thielscher, K. Van Leemput, B. Fischl, A.V. Dalca, J.E. Iglesias
医学图像分析 (2023)
[ 文章 | arxiv | bibtex ]
随后,我们将其扩展到可以处理异构临床扫描,并执行皮质分区和自动质量控制。
用于大规模分析异构临床脑部MRI数据集的鲁棒机器学习分割
B. Billot, M. Colin, Y. Cheng, S.E. Arnold, S. Das, J.E. Iglesias
PNAS (2023)
[ 文章 | arxiv | bibtex ]
在这里,我们发布我们的模型,使用户能够在自己的数据上运行SynthSeg。我们强调,预测结果始终以1mm等方分辨率给出(无论输入分辨率如何)。代码可以在GPU上运行(每次扫描约15秒)或在CPU上运行(约1分钟)。
新特性和更新
2023年3月1日:SynthSeg和SynthSeg 2.0的论文发表了!:open_book: :open_book:
经过SynthSeg(医学图像分析)的长期审核过程,以及SynthSeg 2.0(PNAS)更快的审核过程,两篇论文几乎同时被接受!请参见上面的参考文献或引用部分。
2022年10月4日:SynthSeg现已支持Matlab! :star:
我们很高兴Matlab 2022b(及以后版本)现在在其医学图像工具箱中包含了SynthSeg。他们有一个文档示例说明如何使用它。但为了简化操作,我们编写了自己的Matlab包装器,您可以用一行代码调用它。只需下载这个zip文件,解压缩,打开Matlab,然后输入help SynthSeg获取使用说明。
2022年6月29日:SynthSeg 2.0发布! :v:
除了全脑分割外,它现在还可以进行皮质分区、自动QC和颅内体积(ICV)估计(见下图)。此外,大多数这些功能与SynthSeg 1.0兼容(见表格)。
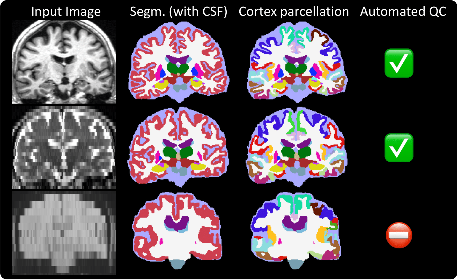

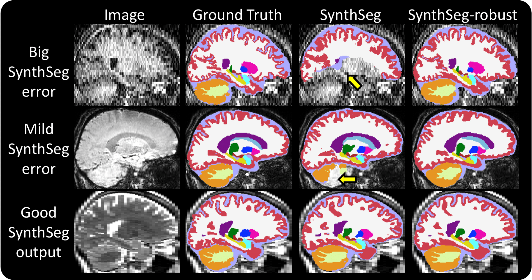
2022年3月1日:鲁棒版本 :hammer:
SynthSeg有时在信噪比低或组织对比度非常低的扫描上会失效。因此,我们开发了一个新模型以提高鲁棒性,称为"SynthSeg-robust"。当SynthSeg给出如下图所示的结果时,您可以使用这种模式:
2021年10月29日:SynthSeg现已在FreeSurfer的开发版本中提供! :tada:
请参见这里了解如何使用它。
一行命令试用!
安装所有Python包后(见下文),您可以简单地使用以下命令在自己的数据上测试SynthSeg:
python ./scripts/commands/SynthSeg_predict.py --i <输入> --o <输出> [--parc --robust --ct --vol <体积> --qc <质量控制> --post <后处理> --resample <重采样>]
其中:
<input>是要分割的扫描或文件夹的路径。也可以是文本文件的路径,每行包含一个要分割的图像路径。<output>是保存输出分割结果的路径。必须与<input>类型相同(即文件路径、文件夹或文本文件,每行包含一个输出分割的路径)。--parc(可选)除全脑分割外还执行皮层区域划分。--robust(可选)使用变体以提高鲁棒性(例如在分析间距较大的临床数据时)。这可能比其他模型更慢。--ct(可选)用于豪斯菲尔德单位的CT扫描。将强度限制在[0, 80]范围内。<vol>(可选)保存所有扫描中所有分割区域体积(单位:mm³)的CSV文件路径(例如 /path/to/volumes.csv)。如果<input>是文本文件,则<vol>也必须是文本文件,每行对应一个受试者的CSV文件路径。<qc>(可选)保存QC分数的CSV文件路径。格式要求与<vol>相同。<post>(可选)保存后验概率软概率图的路径(格式要求与<output>相同)。<resample>(可选)SynthSeg分割结果始终以1mm等方分辨率给出。因此,图像总是在内部重采样到这个分辨率(除非它们已经是1mm分辨率)。使用此标志保存重采样的图像(格式要求与<output>相同)。
还有其他可选标志:
--cpu:(可选)强制代码在CPU上运行,即使有可用的GPU。--threads:(可选)Tensorflow使用的线程数(默认使用一个核心)。使用CPU版本时增加此值可减少运行时间。--crop:(可选)在分割前将输入图像裁剪到给定形状。必须能被32整除。图像围绕中心裁剪,分割结果给出原始大小。可以给出单个整数(如--crop 160)或多个整数(如--crop 160 128 192,按RAS坐标排序)。默认处理整个图像。使用此标志可加快分析或适应GPU内存。--fast:(可选)禁用某些操作以加快预测速度(快一倍,但略微不太准确)。使用 --robust 标志时不适用。--v1:(可选)运行SynthSeg的第一个版本(SynthSeg 1.0,2022年6月29日更新)。
重要提示: SynthSeg始终给出1mm等方分辨率的结果,不受输入影响。但这可能导致某些查看器无法正确将分割结果叠加在相应图像上。在这种情况下,可以使用 --resample 标志获取与分割结果处于相同空间的重采样图像,以便在任何查看器中一起可视化。
完整的分割结构列表及其对应值可在labels table.txt中找到。该表格还详细说明了后验概率图的排序。
安装
-
克隆此仓库。
-
创建虚拟环境(例如使用pip或conda)并安装所有必需的包。 这些依赖于你的Python版本,这里我们列出了Python 3.6(requirements_3.6)和Python 3.8(见requirements_3.8)的要求。 选择权在你,但在每种情况下,请严格遵守确切的包版本。 如果你使用pip,安装依赖项的一种方法是运行setup.py(在激活的虚拟环境中):
python setup.py install。否则,我们还提供了使用pip/conda为Python 3.6/3.8安装所需包的最小命令。
# Conda, Python 3.6:
conda create -n synthseg_36 python=3.6 tensorflow-gpu=2.0.0 keras=2.3.1 h5py==2.10.0 nibabel matplotlib -c anaconda -c conda-forge
# Conda, Python 3.8:
conda create -n synthseg_38 python=3.8 tensorflow-gpu=2.2.0 keras=2.3.1 nibabel matplotlib -c anaconda -c conda-forge
# Pip, Python 3.6:
pip install tensorflow-gpu==2.0.0 keras==2.3.1 nibabel==3.2.2 matplotlib==3.3.4
# Pip, Python 3.8:
pip install tensorflow-gpu==2.2.0 keras==2.3.1 protobuf==3.20.3 numpy==1.23.5 nibabel==5.0.1 matplotlib==3.6.2
-
访问此链接UCL dropbox,下载缺失的模型。然后将它们复制到models文件夹。
-
如果你希望在GPU上运行,还需要安装Cuda(Python 3.6用10.0,Python 3.8用10.1)和CUDNN(两者都用7.6.5)。请注意,如果你使用了conda,这些已经自动安装了。
就是这样!你现在可以使用SynthSeg了!🎉
它是如何工作的?
简而言之,我们使用基于贝叶斯分割前向模型的生成模型,即时采样合成图像来训练网络。关键是,我们采用了领域随机化策略,在每个小批次中从无信息均匀先验中完全随机抽取生成参数。通过向网络展示极其多变的输入数据,我们迫使它学习领域无关的特征。因此,SynthSeg能够直接对任何目标域的真实扫描进�行分割,无需重新训练或微调。
下图首先说明了训练迭代的工作流程,然后概述了生成模型的不同步骤:
[图片:Overview]
最后,我们展示了合成图像的其他示例以及它们的目标分割叠加:
[图片:Training data]
如果您有兴趣了解更多关于SynthSeg的信息,可以阅读相关发表的文章(见下文),并观看这个演讲,该演讲在MIDL 2020上介绍了SynthSeg初步版本的相关文章(对MR对比度而非分辨率的鲁棒性)。
[图片:Talk SynthSeg]
训练您自己的模型
该存储库包含训练、验证和测试您自己的网络所需的所有代码和数据。重要的是,所提出的方法只需要一组解剖分割来进行训练(不需要图像),我们在data中包含了这些数据。虽然提供的函数有详细的文档说明,但我们强烈建议从以下教程开始:
-
1-generation_visualisation:这个非常简单的脚本展示了用于训练SynthSeg的合成图像示例。
-
2-generation_explained:第二个脚本描述了用于控制生成模型的所有参数。我们建议您仔细阅读本教程,因为在开始训练自己的模型之前,了解如何形成合成数据至关重要。
-
3-training:这个脚本重新使用了上一个教程中解释的参数,并专注于学习/架构参数。这里的脚本正是我们用来训练SynthSeg的脚本!
-
4-prediction:这个脚本展示了在网络训练完成后如何进行预测。
-
5-generation_advanced:在这里,我们详细介绍了更高级的生成选项,适用于训练特定对比度和/或分辨率的SynthSeg版本(尽管这些类型的变�体被证明不如在第3个教程中训练的SynthSeg模型表现好)。
-
6-intensity_estimation:这个脚本展示了在训练特定对比度版本的SynthSeg时如何估计GMM的高斯先验。
-
7-synthseg+:最后,我们展示了如何训练SynthSeg的鲁棒版本。
这些教程涵盖了大量材料,将使您能够训练自己的SynthSeg模型。此外,所有函数的文档字符串中提供了更详细的信息,所以不要犹豫查看这些内容!
内容
-
SynthSeg:这是包含生成模型和训练函数的主文件夹:
-
labels_to_image_model.py:包含MRI扫描的生成模型。
-
brain_generator.py:包含
BrainGenerator类,它是labels_to_image_model的包装器。只需实例化该类的对象并调用generate_image()方法即可轻松生成新图像。 -
training.py:包含训练分割网络的代码(解释了所有训练参数)。该函数还展示了如何在训练设置中集成生成模型。
-
predict.py:预测和测试。
-
validate.py:包括验证代码(必须在真实图像上离线进行)。
-
-
models:在这里您可以找到SynthSeg的训练模型。
-
data:如果您想训练自己的SynthSeg模型,该文件夹包含一些大脑标签图示例。
-
script:包含教程以及从终端启动训练和测试的脚本。
-
ext:包括外部包,特别是lab2im包和neuron的修改版本。
引用/联系
此代码采用Apache 2.0许可。
- 如果您使用皮质分区、自动QC或鲁棒版本,请引用以下论文:
用于异质临床脑MRI数据集大规模分析的鲁棒机器学习分割
B. Billot, M. Colin, Y. Cheng, S.E. Arnold, S. Das, J.E. Iglesias
PNAS (2023)
[ 文章 | arxiv | bibtex ]
- 否则,请引用:
SynthSeg:无需重新训练即可对任何对比度和分辨率的脑部MRI扫描进行分割 B. Billot, D.N. Greve, O. Puonti, A. Thielscher, K. Van Leemput, B. Fischl, A.V. Dalca, J.E. Iglesias 医学图像分析 (2023) [ 文章 | arxiv | bibtex ]
如果您对使用此代码有任何疑问,或有任何改进建议,请提出问题或联系我们:bbillot@mit.edu
编辑推荐精选


讯飞智文
一键生成PPT和Word,让学习生活更轻松
讯飞智文是一个利用 AI 技术的项目,能够帮助用户生成 PPT 以及各类文档。无论是商业领域的市场分析报告、年度目标制定,还是学生群体的职业生涯规划、实习避坑指南,亦或是活动策划、旅游攻略等内容,它都能提供支持,帮助用户精准表达,轻松呈现各种信息。


讯飞星火
深度推理能力全新升级,全面对标OpenAI o1
科大讯飞的星火大模型,支持语言理解、知识问答和文本创作等多功能,适用于多种文件和业务场景,提升办公和日常生活的效率。讯飞星火是一个提供丰富智能服务的平台,涵盖科技资讯、图像创作、写作辅助、编程解答、科研文献解读等功能,能为不同需求的用户提供便捷高效的帮助,助力用户轻松获取信息、解决问题,满足多样化使用场景。


Spark-TTS
一种基于大语言模型的高效单流解耦语音令牌文本到语音合成模型
Spark-TTS 是一个基于 PyTorch 的开源文本到语音合成项目,由多个知名机构联合参与。该项目提供了高效的 LLM(大语言模型)驱动的语音合成方案,支持语音克隆和语音创建功能,可通过命令行界面(CLI)和 Web UI 两种方式使用。用户可以根据需求调整语音的性别、音高、速度等参数,生成高质量的语音。该项目适用于多种场景,如有声读物制作、智能语音助手开发等。


Trae
字节跳动发布的AI编程神器IDE
Trae是一种自适应的集成开发环境(IDE),通过自动化和多元协作改变开发流程。利用Trae,团队能够更快速、精确地编写和部署代码,从而提高编程效率和项目交付速度。Trae具备上下文感知和代码自动完成功能,是提升开发效率的理想工具。


咔片PPT
AI助力,做PPT更简单!
咔片是一款轻量化在线演示设计工具,借助 AI 技术,实现从内容生成到智能设计的一站式 PPT 制作服务。支持多种文档格式导入生成 PPT,提供海量模板、智能美化、素材替换等功能,适用于销售、教师、学生等各类人群,能高效制作出高品质 PPT,满足不同场景演示需求。


讯飞绘文
选题、配图、成文,一站式创作,让内容运营更高效
讯飞绘文,一个AI集成平台,支持写作、选题、配图、排版和发布。高效生成适用于各类媒体的定制内容,加速品牌传播,提升内容营销效果。


材料星
专业的AI公文写作平台,公文写作神器
AI 材料星,专业的 AI 公文写作辅助平台,为体制内工作人员提供高效的公文写作解决方案。拥有海量公文文库、9 大核心 AI 功能,支持 30 + 文稿类型生成,助力快速完成领导讲话、工作总结、述职报告等材料,提升办公效率,是体制打工人的得力写作神器。


openai-agents-python
OpenAI Agents SDK,助力开发者便捷使用 OpenAI 相关功能。
openai-agents-python 是 OpenAI 推出的一款强大 Python SDK,它为开发者提供了与 OpenAI 模型交互的高效工具,支持工具调用、结果处理、追踪等功能,涵盖多种应用场景,如研究助手、财务研究等,能显著提升开发效率,让开发者更轻松地利用 OpenAI 的技术优势。


Hunyuan3D-2
高分辨率纹理 3D 资产生成
Hunyuan3D-2 是腾讯开发的用于 3D 资产生成的强大工具,支持从文本描述、单张图片或多视角图片生成 3D 模型,具备快速形状生成能力,可生成带纹理的高质量 3D 模型,适用于多个领域,为 3D 创作提供了高效解决方案。


3FS
一个具备存储、管理和客户端操作等多种功能的分布式文件系统相关项目。
3FS 是一个功能强大的分布式文件系统项目,涵盖了存储引擎、元数据管理、客户端工具等多个模块。它支持多种文件操作,如创建文件和目录、设置布局等,同时具备高效的事件循环、节点选择和协程池管理等特性。适用于需要大规�模数据存储和管理的场景,能够提高系统的性能和可靠性,是分布式存储领域的优质解决方案。
推荐工具精选
AI云服务特惠
懂AI专属折扣关注微信公众号
最新AI工具、AI资讯
独家AI资源、AI项目落地

微信扫一扫关注公众号





