RFDiffusion AA 代码
设置/安装
- 克隆仓库
git clone https://github.com/baker-laboratory/rf_diffusion_all_atom.git
cd rf_diffusion_all_atom
- 下载用于运行RFAA的容器。
wget http://files.ipd.uw.edu/pub/RF-All-Atom/containers/rf_se3_diffusion.sif
- 下载模型权重。
wget http://files.ipd.uw.edu/pub/RF-All-Atom/weights/RFDiffusionAA_paper_weights.pt
- 初始化git子模块
git submodule init
git submodule update
- 安装Apptainer
如果您的系统尚未安装Apptainer,请安装它。这将允许您使用预打包的sif文件运行我们的代码,而无需安装任何Python包:https://apptainer.org/docs/admin/main/installation.html
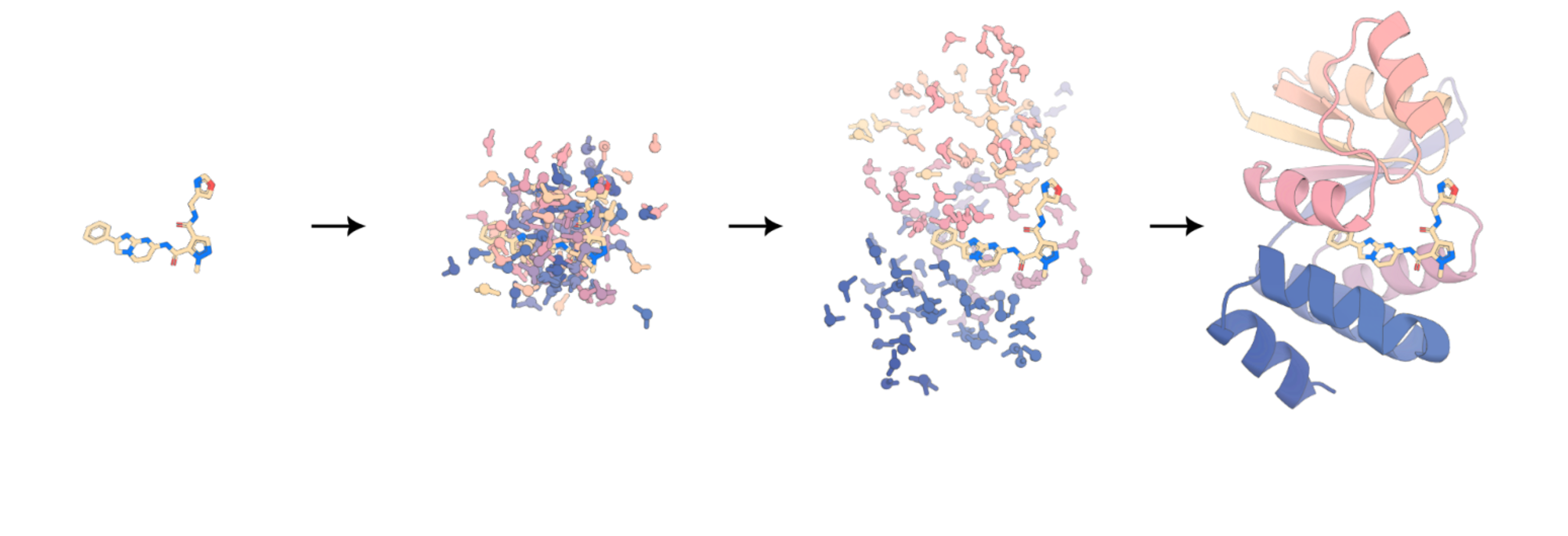
推理
小分子结合蛋白设计
要为PDB 7v11中的配体OQO生成一个结合蛋白,请运行以下命令:
示例(配体结合蛋白):
/usr/bin/apptainer run --nv rf_se3_diffusion.sif -u run_inference.py inference.deterministic=True diffuser.T=100 inference.output_prefix=output/ligand_only/sample inference.input_pdb=input/7v11.pdb contigmap.contigs=[\'150-150\'] inference.ligand=OQO inference.num_designs=1 inference.design_startnum=0
注意:如果不使用GPU,必须省略--nv标志。
参数说明:
inference.deterministic=True为使用的随机数生成器设定种子,以确保结果可重现。即,使用inference.design_startnum=X运行将产生相同的结果。请注意,torch不保证在不同的CPU/GPU架构上的可重现性:https://pytorch.org/docs/stable/notes/randomness.htmlinference.num_designs=1指定将生成1个设计contigmap.contigs=[\'150-150\']指定生成的蛋白质长度应为150diffuser.T=100指定去噪步骤的数量。
预期输出:
output/ligand_only/sample_0.pdb设计的PDB文件output/ligand_only/sample_0_Xt-1_traj.pdb部分去噪的中间结构output/ligand_only/sample_0_X0-1_traj.pdb网络在每一步预测的真实值
请注意,除了给定的基序外,这些结构相关的序列没有任何意义。如果要用于结构预测/表达,必须使用LigandMPNN或类似工具为这些骨架生成序列。
要在基序中包含蛋白质残基A430-435,请使用参数contigmap.contigs。例如,contigmap.contigs=[\'10-120,A84-87,10-120\']告诉模型设计一个包含4个残基基序A84-87的蛋白质,两侧各有10-120个残基。
带蛋白质基序的小分子结合蛋白设计
示例(带蛋白质基序的配体结合蛋白):
/usr/bin/apptainer run --nv rf_se3_diffusion.sif -u run_inference.py inference.deterministic=True diffuser.T=200 inference.output_prefix=output/ligand_protein_motif/sample inference.input_pdb=input/1haz.pdb contigmap.contigs=[\'10-120,A84-87,10-120\'] contigmap.length="150-150" inference.ligand=CYC inference.num_designs=1 inference.design_startnum=0
展示使用RFdiffusionAA、proteinMPNN、AlphaFold2、LigandMPNN和PyRosetta设计血红素结合蛋白的端到端设计流程可在以下链接获取:https://github.com/ikalvet/heme_binder_diffusion

 访问官网
访问官网 Github
Github










