Chroma是一个用于以编程方式设计蛋白质的生成模型。
蛋白质空间复杂且难以驾驭。借助Chroma,蛋白质设计问题可以用可组合的构建模块来表示,从而自动生成多样化的全原子蛋白质结构。作为结构和序列的联合模型,Chroma还可以用于常见的蛋白质建模任务,如根据骨架生成序列、侧链打包和设计评分。
我们提供了各种约束条件的蛋白质条件约束,包括子结构、对称性、形状以及某些蛋白质类别和注释的神经网络预测。我们还提供了一个API,可以让你用几行代码创建自己的条件约束。
在内部,Chroma使用扩散建模、等变图神经网络和条件随机场,以亚平方复杂度高效地对全原子结构进行采样。它可以在普通GPU上几分钟内生成大型复合物。你可以在我们的论文用可编程生成模型照亮蛋白质空间(Nature 2023)中了解更多关于Chroma的信息,包括一些早期设计的生物物理和晶体学验证。
开始使用
**注意:**下载和使用预训练模型权重需要API密钥。可以在这里获取。
Colab笔记本。开始使用Chroma最快的方法是我们的Colab笔记本,它在预配置的浏览器环境中为各种用例提供了起点
- Chroma快速入门:演示使用Chroma无条件和有条件生成蛋白质的GUI笔记本。
- Chroma API教程:直接在
python中演示蛋白质I/O、采样和设计配置的代码笔记本。 - Chroma条件约束API教程:深入探讨实现新的Chroma条件约束的内部原理。
PyPi包。你可以使用以下命令安装最新版本的Chroma:
pip install generate-chroma
从github安装最新的Chroma
git clone https://github.com/generatebio/chroma.git
pip install -e chroma # 使用`-e`可在本地进行编辑。
采样
无条件单体。我们为无条件和有条件蛋白质设计提供了一个统一的入口点,即Chroma.sample()方法。当没有指定条件约束时,我们可以使用以下代码采样一个简单的200个氨基酸的单体蛋白:
from chroma import Chroma
chroma = Chroma()
protein = chroma.sample(chain_lengths=[200])
protein.to("sample.cif")
display(protein)
通常,Chroma.sample()接受设计超参数和条件约束作为输入,并输出表示蛋白质系统全原子结构的Protein对象,这些对象可以以PDB或mmCIF格式从磁盘加载和保存。
无条件复合物。要采样复合物而不是单体,我们只需要:
from chroma import Chroma
chroma = Chroma()
protein = chroma.sample(chain_lengths=[100, 200])
protein.to("sample-complex.cif")
有条件复合物。我们可以通过条件约束和采样超参数进一步定制采样以达到设计目标。例如,要采样一个每个单体有100个残基的C3对称同源三聚体,我们可以:
from chroma import Chroma, conditioners
chroma = Chroma()
conditioner = conditioners.SymmetryConditioner(G="C_3", num_chain_neighbors=2)
protein = chroma.sample(
chain_lengths=[100],
conditioner=conditioner,
langevin_factor=8,
inverse_temperature=8,
sde_func="langevin",
potts_symmetry_order=conditioner.potts_symmetry_order)
protein.to("sample-C3.cif")
由于条件约束的组合本身也是条件约束,即使是相对复杂的设计问题也可以遵循这种基本的使用模式。有关超参数、条件约束和起点的更多信息,请参阅演示笔记本和文档字符串。
设计
稳健设计。Chroma是序列和结构的联合模型,使用共同的图神经网络基础架构来参数化骨架生成和条件序列及侧链生成。这些序列和侧链解码器是扩散感知的,意味着它们不仅被训练来预测扩散时间$t=0$时的自然结构的序列和侧链,还被训练来预测所有扩散时间$t \in [0,1]$上的噪声结构。因此,设计网络的$t$超参数通过扩散增强提供了一种可调节的稳健性,我们在模型尝试设计精确指定的骨架(例如$t=0.0$)和在附近骨架构象的小邻域内进行稳健设计(例如$t=0.5$)之间进行权衡。
虽然Chroma论文中呈现的所有结果都是在$t=0.0$时进行的精确设计,但我们发现在接近$t=0.5$时进行稳健设计经常可以改善一次性重折叠,同时只对目标骨架产生微小的、通常是埃级的弛豫调整。当我们比较这两种设计模式在论文中分析的50,000个无条件骨架集上的表现时,我们看到AlphaFold和ESMFold的重折叠性能都有很大改善,这种改善在蛋白质长度、螺旋比例或与已知结构的相似性方面都有很好的分层(参见Chroma补充图14以获取更多背景信息)。
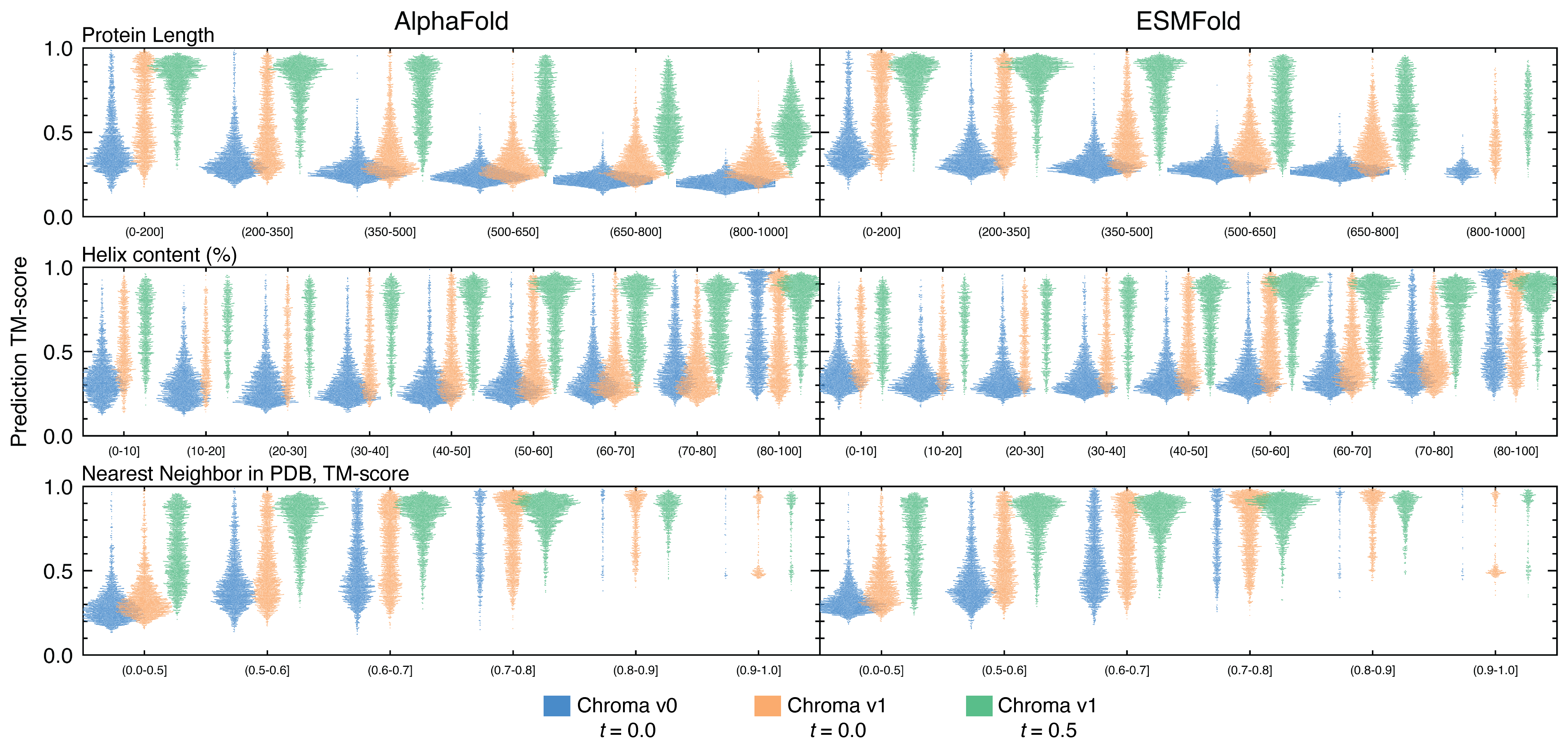
扩散时间条件 $t$ 的值可以通过 Chroma.sample 和 Chroma.design 中的 design_t 参数设置。我们发现对于生成的结构,$t = 0.5$ 可以产生高度稳健的重折叠结果,因此是默认设置。对于实验精确的结构,$t = 0.0$ 可能更合适,而介于两者之间的值可能在这两种情况之间提供有用的权衡。
自由设计。可以单独访问 Chroma 的设计网络来设计、重新设计和打包任意蛋白质系统。这里我们从 PDB 加载一个蛋白质并重新设计如下:
# 重新设计一个蛋白质
from chroma import Protein, Chroma
chroma = Chroma()
protein = Protein('1GFP')
protein = chroma.design(protein)
protein.to("1GFP-redesign.cif")
子序列重新设计也可用,并与内置的选择代数兼容,同时还可以使用特定位置和突变的掩码约束,如下所示:
# 重新设计一个蛋白质
from chroma import Protein, Chroma
chroma = Chroma()
protein = Protein('my_favorite_protein.cif') # PDB 格式也可以
protein = chroma.design(protein, design_selection="resid 20-50 around 5.0") # 围绕索引 20-50 的 5 埃半径范围
protein.to("my_favorite_protein_redesign.cif")
我们在演示笔记本中提供了更多设计示例。
条件器
使用 Chroma 进行蛋白质设计是可编程的。我们的 Conditioner 框架允许在任意组合的蛋白质规格下进行自动条件采样,这些规格可以是约束(偏置状态分布)或限制(直接限制底层采样过程的域);参见我们的论文中的补充附录 M。我们预定义了多个条件器,包括用于控制子结构、对称性、形状、语义和自然语言提示的条件器(见 chroma.layers.structure.conditioners),这些可以任意组合使用。
| 条件器 | chroma.conditioners 中的类 | 应用示例 |
|---|---|---|
| 对称性约束 | SymmetryConditioner, ScrewConditioner | 大型对称组装体 |
| 子结构约束 | SubstructureConditioner | 子结构嫁接,骨架强制 |
| 形状约束 | ShapeConditioner | 分子形状控制 |
| 二级结构 | ProClassConditioner | 二级结构规定 |
| 域分类 | ProClassConditioner | 类别规定,如 Pfam、CATH 或分类学 |
| 文本说明 | ProCapConditioner | 自然语言提示 |
| 序列 | SubsequenceConditioner | 子序列约束 |
工作原理。条件器的核心思想是可组合的状态转换,其中每个条件器都是一个以可微分方式修改蛋白质系统状态和/或能量的函数(补充附录 M)。例如,要将对称性编码为约束,我们可以将不对称单元作为输入,并根据所需的对称群对其进行镶嵌,从而输出一个在结构上对称的蛋白质系统。对于像神经网络约束这样的东西,我们可以通过目标条件的负对数概率来调整总系统能量。对于这两种情况,我们将扩散能量添加到条件器的输出中,然后通过所有中间转换反向传播总能量,以计算与通用采样 SDE(如退火 Langevin 动力学)兼容的无约束力。
我们在下面概述了这个整体条件器框架。
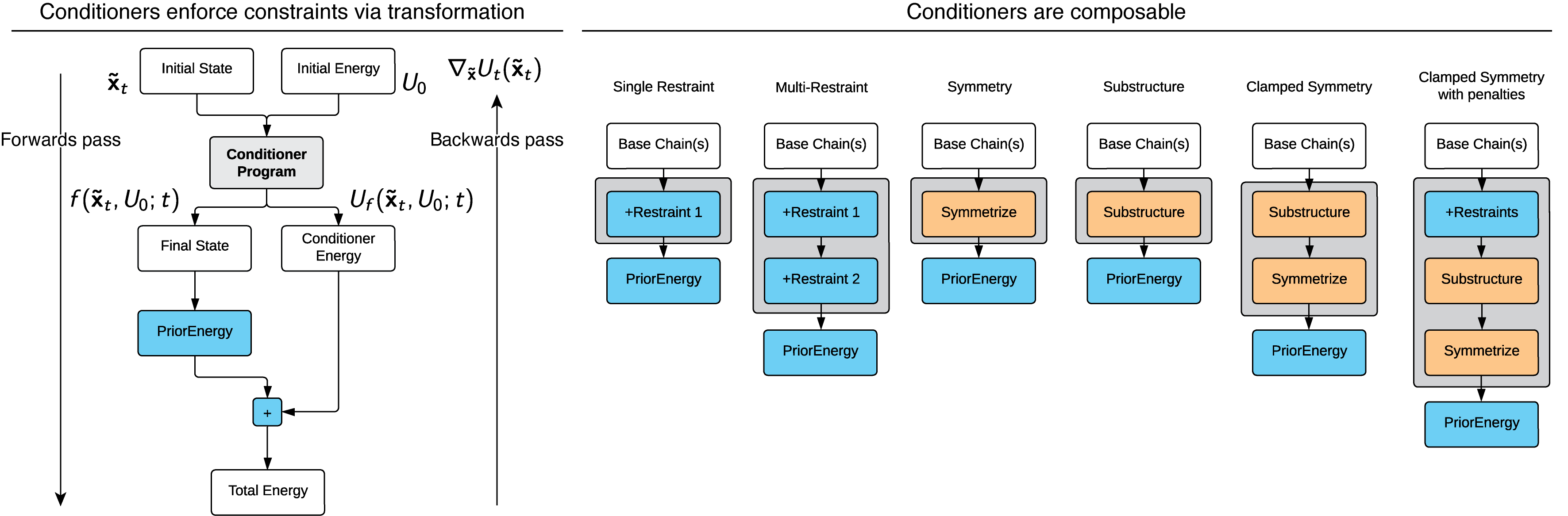
Conditioner 类是 Chroma 蛋白质设计的可组合构建块。条件器 API
开发新的条件器很简单。Conditioner 是一个 Pytorch nn.Module,它接收系统状态 - 即结构、能量和扩散时间 - 并输出可能更新的结构和能量,如下所示:
class Conditioner(torch.nn.Module):
"""用于参数化蛋白质设计问题的可组合函数。
"""
def __init__(self, *args, **kwargs):
super().__init__()
# 设置条件器的超参数
def forward(
self,
X: torch.Tensor, # 输入坐标
C: torch.LongTensor, # 输入链图(用于复合体)
O: torch.Tensor, # 输入序列(one-hot,未使用)
U: torch.Tensor, # 输入能量(one-hot,未使用)
t: Union[torch.Tensor, float], # 扩散时间
):
# 更新状态,例如从无约束映射到有约束的流形
X_update, C_update = update_state(X, C, t)
# 更新能量,例如添加约束势能
U_update = U + update_energy(X, C, t)
return X_update, C_update, O, U_update, t
大致来说,Conditioner 在结构上是可组合的,因为它们的输入和输出类型签名是匹配的(即它们是自同态)。因此,我们也可以像开发传统神经网络层一样,简单地通过"堆叠"条件器来构建条件器。使用最终的 Conditioner 作为输入,Chroma.sample() 将利用 Pytorch 的自动微分功能自动提供一个扩散退火的 MCMC 采样算法来使用这个条件器进行采样(我们注意到这并不是魔法,适当地缩放和参数化很重要)。
最小条件器:2D 晶格对称性
下面的代码片段展示了如何用几行代码添加一个条件器,该条件器规定生成一个 2D 晶体状对象,其中生成的蛋白质排列在 M x N 的矩形晶格中。
import torch
from chroma.models import Chroma
from chroma.layers.structure import conditioners
class Lattice2DConditioner(conditioners.Conditioner):
def __init__(self, M, N, cell):
super().__init__()
# 设置二维格子的坐标
self.order = M*N
x = torch.arange(M) * cell[0]
y = torch.arange(N) * cell[1]
xx, yy = torch.meshgrid(x, y, indexing="ij")
dX = torch.stack([xx.flatten(), yy.flatten(), torch.zeros(M * N)], dim=1)
self.register_buffer("dX", dX)
def forward(self, X, C, O, U, t):
# 在格子上平铺单位晶胞
X = (X[:,None,...] + self.dX[None,:,None,None]).reshape(1, -1, 4, 3)
C = torch.cat([C + C.unique().max() * i for i in range(self.dX.shape[0])], dim=1)
# 对梯度进行分组平均(简化力的缩放)
X.register_hook(lambda gradX: gradX / self.order)
return X, C, O, U, t
chroma = Chroma().cuda()
conditioner = Lattice2DConditioner(M=3, N=4, cell=[20., 15.]).cuda()
protein = chroma.sample(
chain_lengths=[70], conditioner=conditioner, sde_func='langevin',
potts_symmetry_order=conditioner.order
)
protein.to_CIF("lattice_protein.cif")

关于条件约束器的说明
这个条件约束器框架的一个吸引人的方面是它非常通用,既可以实现约束(涉及对x的操作),也可以实现限制(相当于对U的更改)。同时,在限制条件下的生成仍然可能(而且经常)具有挑战性,因为由此产生的有效能量景观可能变得任意崎岖难以积分。因此,我们建议在使用和开发新的条件约束器或条件约束器组合时要谨慎。我们发现检查扩散轨迹(包括无约束和去噪轨迹,$\hat{x}_t$和$\tilde{x}_t$)可以作为识别积分挑战和定义更好的条件约束器形式或更好的采样方案的有效工具。
引用Chroma
如果您在研究中使用了Chroma,请引用:
J. B. Ingraham, M. Baranov, Z. Costello, K. W. Barber, W. Wang, A. Ismail, V. Frappier, D. M. Lord, C. Ng-Thow-Hing, E. R. Van Vlack, S. Tie, V. Xue, S. C. Cowles, A. Leung, J. V. Rodrigues, C. L. Morales-Perez, A. M. Ayoub, R. Green, K. Puentes, F. Oplinger, N. V. Panwar, F. Obermeyer, A. R. Root, A. L. Beam, F. J. Poelwijk, and G. Grigoryan, "Illuminating protein space with a programmable generative model", Nature, 2023 (10.1038/s41586-023-06728-8).
@Article{Chroma2023,
author = {Ingraham, John B. and Baranov, Max and Costello, Zak and Barber, Karl W. and Wang, Wujie and Ismail, Ahmed and Frappier, Vincent and Lord, Dana M. and Ng-Thow-Hing, Christopher and Van Vlack, Erik R. and Tie, Shan and Xue, Vincent and Cowles, Sarah C. and Leung, Alan and Rodrigues, Jo\~{a}o V. and Morales-Perez, Claudio L. and Ayoub, Alex M. and Green, Robin and Puentes, Katherine and Oplinger, Frank and Panwar, Nishant V. and Obermeyer, Fritz and Root, Adam R. and Beam, Andrew L. and Poelwijk, Frank J. and Grigoryan, Gevorg},
journal = {Nature},
title = {Illuminating protein space with a programmable generative model},
year = {2023},
volume = {},
number = {},
pages = {},
doi = {10.1038/s41586-023-06728-8}
}
致谢
Chroma代码库是Generate Biomedicines公司许多贡献者的工作成果。我们要感谢:Ahmed Ismail, Alan Witmer, Alex Ramos, Alexander Bock, Ameya Harmalkar, Brinda Monian, Craig Mackenzie, Dan Luu, David Moore, Frank Oplinger, Fritz Obermeyer, George Kent-Scheller, Gevorg Grigoryan, Jacob Feala, James Lucas, Jenhan Tao, John Ingraham, Martin Jankowiak, Max Baranov, Meghan Franklin, Mick Ward, Rudraksh Tuwani, Ryan Nelson, Shan Tie, Vincent Frappier, Vincent Xue, William Wolfe-McGuire, Wujie Wang, Zak Costello, Zander Harteveld。
许可证
版权所有 Generate Biomedicines, Inc.
Chroma代码许可
根据Apache许可证2.0版("许可证")授权;除非遵守许可证,否则您不得使用此代码。您可以在https://www.apache.org/licenses/LICENSE-2.0获得许可证的副本。
除非适用法律要求或书面同意,否则根据许可证分发的软件是基于"按原样"的基础分发的,不附带任何明示或暗示的担保或条件,包括但不限于对所有权、非侵权、适销性或特定用途适用性的任何担保或条件。有关许可证下权限和限制的具体规定,请参阅许可证。
模型权重许可
Chroma权重可供接受并同意受Chroma参数许可条款约束的学术研究人员和非营利实体免费使用。请访问权重下载页面了解更多信息。如果您不符合根据提供的许可使用Chroma参数的资格,或者如果您希望在许可授予的权利范围之外共享和/或使用Chroma参数(包括用于商业目的),您可以通过以下方式联系许可方:licensing@generatebiomedicines.com。

 访问官网
访问官网 Github
Github Huggingface
Huggingface 论文
论文










