ESM3
ESM3是一个前沿的生物学生成模型,能够同时对蛋白质的三个基本生物学特性进行推理:序列、结构和功能。这三种数据模态在ESM3的输入和输出中以离散令牌的轨道形式表示。您可以向模型呈现跨轨道的部分输入组合,ESM3将为所有轨道提供输出预测。
ESM3是一个生成式掩码语言模型。您可以用部分序列、结构和功能关键词来提示它,并迭代采样被掩码的位置,直到所有位置都被解码。这种迭代采样就是.generate()函数所做的事情。
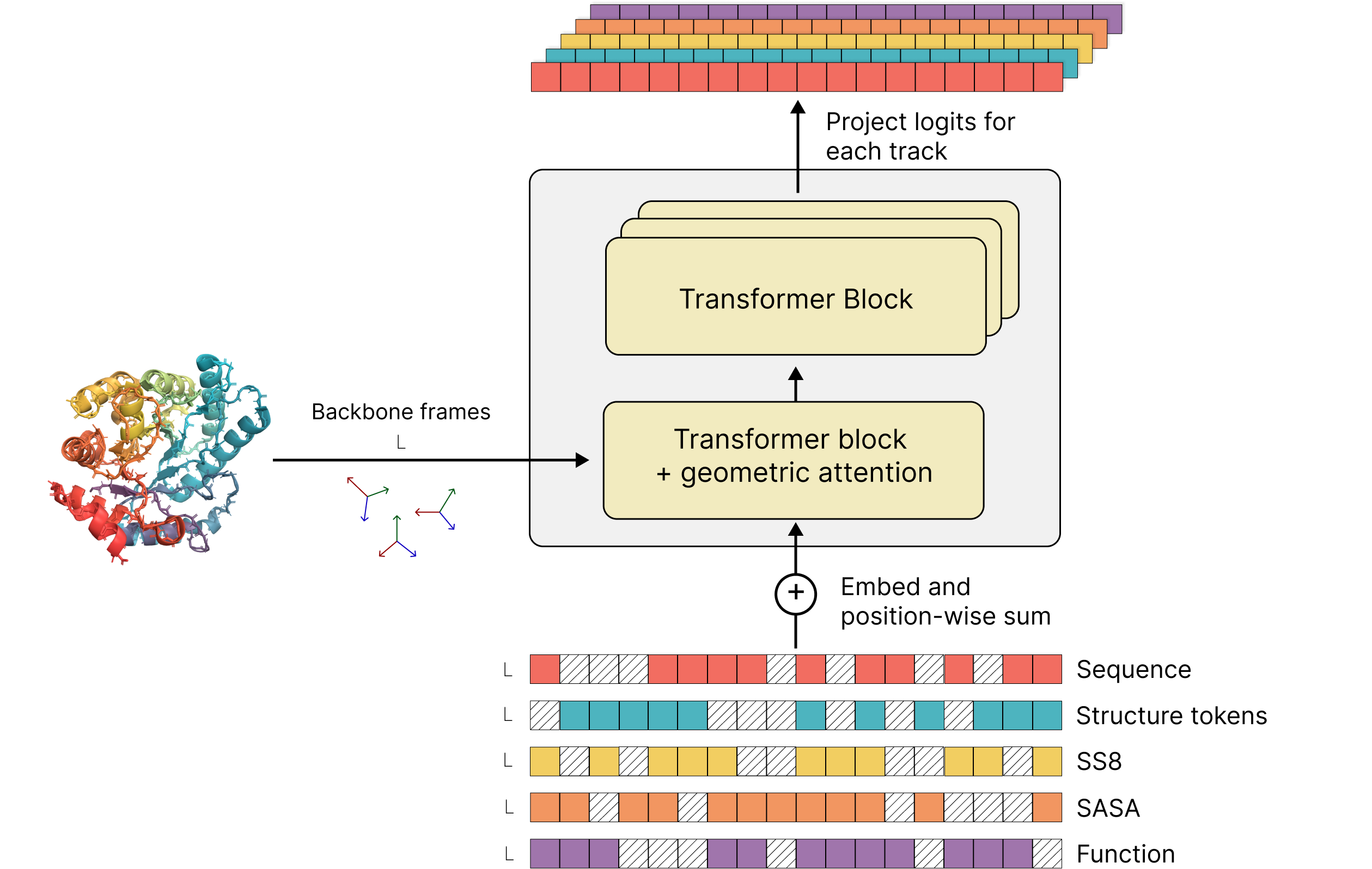
由于其transformer骨干和对离散令牌序列的全对全推理,ESM3架构具有高度可扩展性。在其最大规模时,ESM3在2.78亿个蛋白质和7710亿个唯一令牌上进行了1.07e24次浮点运算的训练,拥有980亿个参数。 通过阅读博客文章和预印本(Hayes等人,2024)了解更多信息。
这里我们介绍esm3-open-small。它拥有14亿个参数,是该系列中最小和最快的模型。
ESM3-open在非商业许可下可用,许可内容可在LICENSE.md中查看。
访问我们的讨论页面以取得联系、提供反馈、提问或分享您使用ESM3的经验!
ESM3-open快速入门
pip install esm
为了下载权重,我们要求用户接受我们的非商业许可。 权重存储在HuggingFace Hub上的HuggingFace/EvolutionaryScale/esm3。 请创建一个账户并接受许可。
from huggingface_hub import login
from esm.models.esm3 import ESM3
from esm.sdk.api import ESM3InferenceClient, ESMProtein, GenerationConfig
# 将指导您如何从huggingface hub获取API密钥,创建一个具有"Read"权限的密钥。
login()
# 这将下载模型权重并在您的机器上实例化模型。
model: ESM3InferenceClient = ESM3.from_pretrained("esm3_sm_open_v1").to("cuda") # 或 "cpu"
# 为部分碳酸酐酶(2vvb)生成一个补全
prompt = "___________________________________________________DQATSLRILNNGHAFNVEFDDSQDKAVLKGGPLDGTYRLIQFHFHWGSLDGQGSEHTVDKKKYAAELHLVHWNTKYGDFGKAVQQPDGLAVLGIFLKVGSAKPGLQKVVDVLDSIKTKGKSADFTNFDPRGLLPESLDYWTYPGSLTTPP___________________________________________________________"
protein = ESMProtein(sequence=prompt)
# 先生成序列,然后生成结构。这将迭代解码序列轨道。
protein = model.generate(protein, GenerationConfig(track="sequence", num_steps=8, temperature=0.7))
# 我们可以展示生成序列的预测结构。
protein = model.generate(protein, GenerationConfig(track="structure", num_steps=8))
protein.to_pdb("./generation.pdb")
# 然后我们可以通过反向折叠序列并重新计算结构来进行往返设计
protein.sequence = None
protein = model.generate(protein, GenerationConfig(track="sequence", num_steps=8))
protein.coordinates = None
protein = model.generate(protein, GenerationConfig(track="structure", num_steps=8))
protein.to_pdb("./round_tripped.pdb")
恭喜您刚刚使用ESM3生成了第一批蛋白质! 让我们在笔记本和脚本的帮助下探索一些更高级的提示技巧。
generate.ipynb将通过两个提示示例(支架搭建和二级结构编辑)使用开放模型进行演示:
gfp_design.ipynb将演示我们用于设计esmGFP的更复杂的生成过程:
我们还在examples/下提供了展示常见工作流程的示例脚本:
- local_generate.py展示了常见任务的简单优雅之处:它通过仅调用
model.generate()进行迭代解码,展示了折叠、反向折叠和思维链生成。 - seqfun_struct.py展示了如何将模型作为标准PyTorch模型直接使用,只需简单调用模型的
forward函数。
Forge:访问更大的ESM3模型
您可以在EvolutionaryScale Forge申请beta访问完整系列的更大和更高能力的ESM3模型。
我们鼓励用户通过Python esm库而不是命令行与Forge API进行交互。
Python接口使您能够交互式地加载蛋白质、构建提示,并使用ESMProtein和配置类检查生成的蛋白质,这些类也用于与本地模型交互。
在任何示例脚本中,尝试用Forge API客户端替换本地ESM3模型:
# 不在本地机器上加载模型:
model: ESM3InferenceClient = ESM3.from_pretrained("esm3_sm_open_v1").to("cuda") # 或 "cpu"
# 只需将该行替换为:
model: ESM3InferenceClient = esm.sdk.client("esm3-md-v1", token="<your forge token>")
# 现在您就在与在我们远程服务器上运行的模型进行交互。
...
完全相同的代码将继续工作。 这使得从较小且更快的模型无缝过渡到我们用于蛋白质设计工作的980亿参数的大型蛋白质语言模型成为可能。
负责任的开发
EvolutionaryScale是一家公益公司。我们的使命是通过与科学界合作,以及开放、安全和负责任的研究,开发人工智能来理解生物学,造福人类健康和社会。受到我们领域历史以及新原则和建议的启发,我们创建了一个负责任的开发框架,以透明和清晰的方式指导我们朝着使命前进。
我们框架的核心原则是:
- 我们将传达我们研究的益处和风险
- 我们将在公开部署之前主动和严格地评估我们模型的风险
- 我们将采用风险缓解策略和预防性保障措施
- 我们将与政府、政策和公民社会的利益相关者合作,使他们保持知情
考虑到这一点,我们对esm3-sm-open-v1进行了各种缓解措施,详情见我们的论文
许可证
总体情况:
-
EvolutionaryScale AI模型仅根据本社区许可协议对个人或非商业组织(包括大学、非营利组织和研究机构、教育和政府机构)提供非商业用途。
-
您不得将EvolutionaryScale AI模型或其任何衍生作品或输出用于:
-
任何商业活动,例如,任何由商业实体、代表商业实体或为商业实体进行的活动,或开发任何产品或服务,如在API后托管AI模型;或
-
在未注明EvolutionaryScale和本社区许可协议的情况下使用;或
-
-
禁止训练与进化尺度人工智能模型相似的人工智能驱动的第三方模型,即使是非商业用途。但是,您可以创建ESM3的衍生作品,例如通过微调或添加模型层。
-
您可以根据社区许可协议发布、分享和改编进化尺度人工智能模型及其输出,用于非商业目的,包括对改编模型施加非商业限制。
在使用ESM3之前,请阅读我们的非商业社区许可协议,该协议可在./LICENSE.md下找到。

 访问官网
访问官网 Github
Github Huggingface
Huggingface 论文
论文










