SAMed
本仓库包含以下论文的实现:
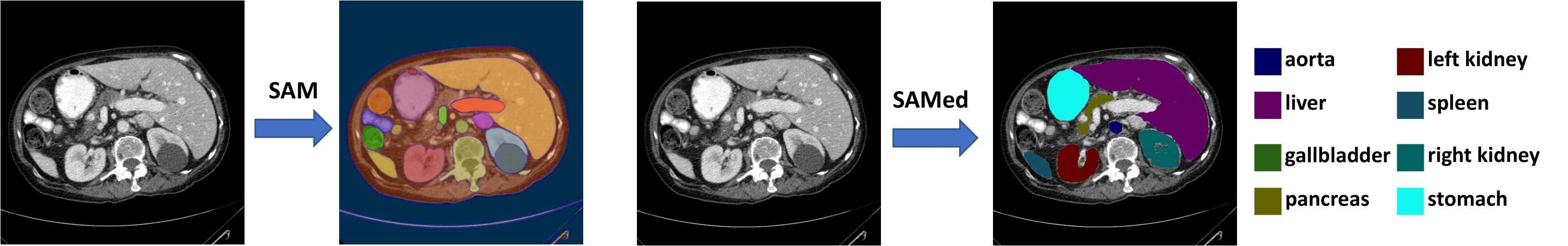
:star: 新闻
- 感谢我的导师的大力投入,我得以微调SAM的
vit_h版本,以实现更精确的医学图像分割。现在,我们发布了SAMed的vit_h版本(我们将此版本称为SAMed_h),下表展示了SAMed和SAMed_h的对比。
| 模型 | DSC | HD | 主动脉 | 胆囊 | 左肾 | 右肾 | 肝脏 | 胰腺 | 脾脏 | 胃 |
|---|---|---|---|---|---|---|---|---|---|---|
| SAMed | 81.88 | 20.64 | 87.77 | 69.11 | 80.45 | 79.95 | 94.80 | 72.17 | 88.72 | 82.06 |
| SAMed_h | 84.30 | 16.02 | 87.81 | 74.72 | 85.76 | 81.52 | 95.76 | 70.63 | 90.46 | 87.77 |
无需任何技巧,SAMed_h的性能就大幅超越了SAMed。尽管vit_h版本的模型大小比vit_b版本大得多(超过2G,而vit_b约350M),但SAMed_h的LoRA检查点并没有增加很多(从18M增加到21M)。因此,SAMed_h的部署和存储成本与SAMed几乎相当。鉴于业界倾向于部署更大、性能更好的模型,我们相信SAMed_h在实际的计算机辅助诊断和术前规划中更有前景。有关SAMed_h的更多细节,请访问SAMed_h目录。
概述
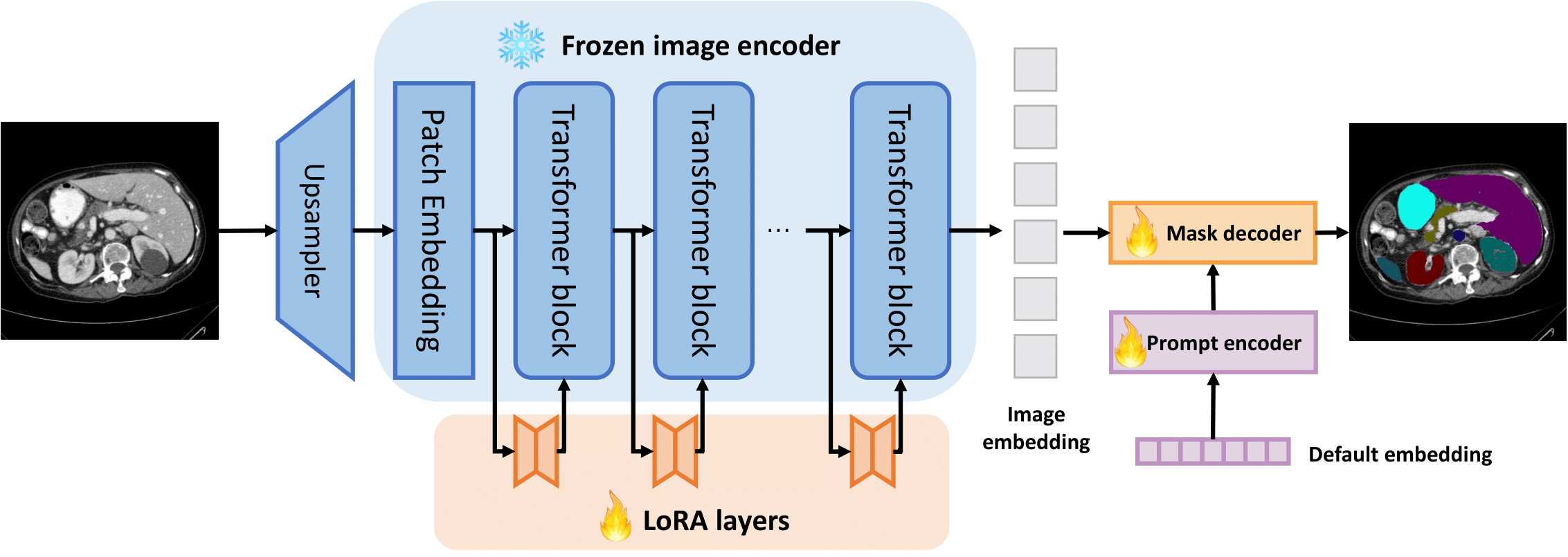 我们提出了SAMed,一个用于医学图像分割的通用解决方案。与之前的方法不同,SAMed基于大规模图像分割模型Segment Anything Model (SAM)构建,探索了为医学图像分割定制大规模模型的新研究范式。SAMed采用基于低秩(LoRA)的微调策略来微调SAM的图像编码器,并将其与提示编码器和掩码解码器一起在标注的医学图像分割数据集上进行微调。我们还观察到预热微调策略和AdamW优化器能够使SAMed成功收敛并获得更低的损失。与SAM不同,SAMed可以对医学图像进行语义分割。我们训练的SAMed模型在Synapse多器官分割数据集上达到了81.88的DSC和20.64的HD,与最先进的方法相当。我们进行了广泛的实验来验证我们设计的有效性。由于SAMed只更新了SAM参数的一小部分,其部署成本和存储成本在实际使用中相当低。
我们提出了SAMed,一个用于医学图像分割的通用解决方案。与之前的方法不同,SAMed基于大规模图像分割模型Segment Anything Model (SAM)构建,探索了为医学图像分割定制大规模模型的新研究范式。SAMed采用基于低秩(LoRA)的微调策略来微调SAM的图像编码器,并将其与提示编码器和掩码解码器一起在标注的医学图像分割数据集上进行微调。我们还观察到预热微调策略和AdamW优化器能够使SAMed成功收敛并获得更低的损失。与SAM不同,SAMed可以对医学图像进行语义分割。我们训练的SAMed模型在Synapse多器官分割数据集上达到了81.88的DSC和20.64的HD,与最先进的方法相当。我们进行了广泛的实验来验证我们设计的有效性。由于SAMed只更新了SAM参数的一小部分,其部署成本和存储成本在实际使用中相当低。
待办事项
- 制作演示。
- 在更多数据集上微调
-
基于SAM的vit_l或vit_h模式制作SAMed
先决条件
- Linux (我们在Ubuntu 18.04上测试了我们的代码)
- Anaconda
- Python 3.7.11
- Pytorch 1.9.1
首先,请克隆仓库
git clone https://github.com/hitachinsk/SAMed.git
然后,请运行以下命令:
conda create -n SAMed python=3.7.11
conda activate SAMed
pip install -r requirements.txt
如果您有原始的Synapse数据集,我们提供了预处理脚本来处理和规范化用于训练的数据。请参考该文件夹了解更多详情。
快速开始
目前,我们提供了SAMed和SAMed_s模型以便快速复现我们的结果。LoRA检查点及其对应的配置如下表所示。
以下是使用说明:
- 切换目录到此仓库的根目录。
- 请下载预训练的SAM模型(由SAM的原始仓库提供)和SAMed的LoRA检查点。将它们放在
./checkpoints文件夹中。 - 请下载测试集并将其放在./testset文件夹中。然后解压并删除该文件。
- 运行以下命令来测试SAMed的性能。
python test.py --is_savenii --output_dir <您的输出目录>
如果一切正常,您会发现平均DSC为0.8188(81.88),HD为20.64,这与论文中的表1相对应。并在<您的输出目录>中查看测试结果。
此外,我们还提供了SAMed_s模型,它使用LoRA对图像编码器和掩码解码器中的transformer块进行微调。与SAMed相比,SAMed_s的模型尺寸更小,但性能也略有下降。如果您想使用这个模型,请下载并将其放在./checkpoints_s文件夹中,然后运行以下命令来测试其性能。
python test.py --is_savenii --output_dir <您的输出目录> --lora_ckpt checkpoints_s/epoch_159.pth --module sam_lora_image_encoder_mask_decoder
SAMed_s的平均DSC为0.7778(77.78),HD为31.72,这与论文中的表3相对应。
训练
我们使用2个RTX 3090 GPU进行训练。
- 请下载经过处理的训练集,其分辨率为
224x224,并将其放在<您的文件夹>中。然后解压并删除该文件。我们还准备了分辨率为512x512的训练集供参考,224x224版本的训练集是从512x512版本下采样得到的。 - 运行以下命令来训练SAMed。
python train.py --root_path <您的文件夹> --output <您的输出路径> --warmup --AdamW
在<您的输出路径>中查看结果。
许可证
本工作采用MIT许可证。详情请参见LICENSE。
引用
如果我们的工作启发了您的研究,或者代码的某些部分对您的工作有用,请引用我们的论文:
@article{samed,
title={Customized Segment Anything Model for Medical Image Segmentation},
author={Kaidong Zhang, and Dong Liu},
journal={arXiv preprint arXiv:2304.13785},
year={2023}
}
联系方式
如果您有任何问题,请通过以下方式联系我们:
致谢
我们感谢Segment Anything Model的开发者和Synapse多器官分割数据集的提供者。SAMed的代码基于TransUnet和SAM LoRA构建,我们对这些优秀的项目表示感谢。

 Github
Github 文档
文档 论文
论文










