GenSLMs:全基因组语言模型揭示SARS-CoV-2进化动态
预印本
可在此处获取:https://www.biorxiv.org/content/10.1101/2022.10.10.511571v2
目录
安装
在大多数系统上安装genslm:
pip install git+https://github.com/ramanathanlab/genslm
GenSLMs在Polaris和Perlmutter超级计算机上进行训练。有关这些系统上的安装,请参阅INSTALL.md。
使用
:warning: 模型权重将在2023年5月5日至2023年5月12日期间不可用
:warning: 2023年5月3日之前下载的模型权重在命名空间上存在小问题。请重新下载模型以修复。
我们的预训练模型和数据集可以从这个Globus端点下载。
使用GenSLMs计算序列嵌入用于下游任务,生成合成序列,或轻松扩展到您自己的应用程序。
计算嵌入 
import torch
import numpy as np
from torch.utils.data import DataLoader
from genslm import GenSLM, SequenceDataset
# 加载模型
model = GenSLM("genslm_25M_patric", model_cache_dir="/content/gdrive/MyDrive")
model.eval()
# 如果有GPU则选择GPU设备,否则使用CPU
device = "cuda" if torch.cuda.is_available() else "cpu"
model.to(device)
# 输入数据是一个基因序列列表
sequences = [
"ATGAAAGTAACCGTTGTTGGAGCAGGTGCAGTTGGTGCAAGTTGCGCAGAATATATTGCA",
"ATTAAAGATTTCGCATCTGAAGTTGTTTTGTTAGACATTAAAGAAGGTTATGCCGAAGGT",
]
dataset = SequenceDataset(sequences, model.seq_length, model.tokenizer)
dataloader = DataLoader(dataset)
# 计算每个输入序列的平均嵌入
embeddings = []
with torch.no_grad():
for batch in dataloader:
outputs = model(
batch["input_ids"].to(device),
batch["attention_mask"].to(device),
output_hidden_states=True,
)
# outputs.hidden_states形状:(layers, batch_size, sequence_length, hidden_size)
# 使用最后一层的嵌入
emb = outputs.hidden_states[-1].detach().cpu().numpy()
# 计算序列长度的平均值
emb = np.mean(emb, axis=1)
embeddings.append(emb)
# 将嵌入连接成形状为(num_sequences, hidden_size)的数组
embeddings = np.concatenate(embeddings)
embeddings.shape
>>> (2, 512)
生成合成序列 
from genslm import GenSLM
# 加载模型
model = GenSLM("genslm_25M_patric", model_cache_dir="/content/gdrive/MyDrive")
model.eval()
# 如果有GPU则选择GPU设备,否则使用CPU
device = "cuda" if torch.cuda.is_available() else "cpu"
model.to(device)
# 用起始密码子提示语言模型
prompt = model.tokenizer.encode("ATG", return_tensors="pt").to(device)
tokens = model.model.generate(
prompt,
max_length=10, # 增加此值以生成更长的序列
min_length=10,
do_sample=True,
top_k=50,
top_p=0.95,
num_return_sequences=2, # 更改要生成的序列数量
remove_invalid_values=True,
use_cache=True,
pad_token_id=model.tokenizer.encode("[PAD]")[0],
temperature=1.0,
)
sequences = model.tokenizer.batch_decode(tokens, skip_special_tokens=True)
for sequence in sequences:
print(sequence)
>>> ATG GTT ATT TCA TCT GAT TTA CCA ACT
>>> ATG TTC ATT CTT CCG GCA CTT ATC GAA
扩散模型
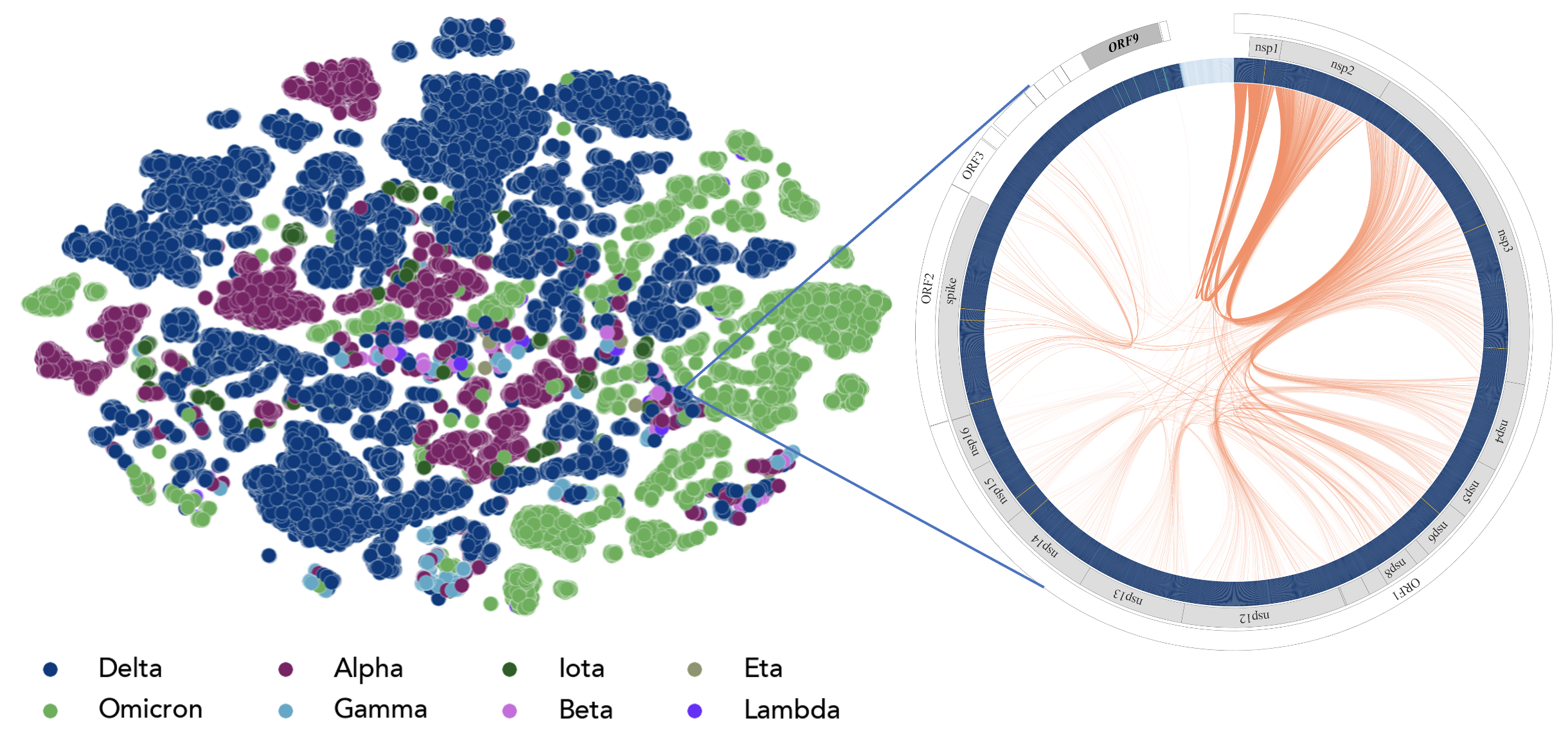
一种新颖的具有两个层次的分层语言模型:顶层使用扩散模型来捕获全局上下文和整个基因组序列的长程相互作用;底层使用transformer进行密码子级建模,由顶层扩散模型指导。该模型通过利用其生成能力,使我们能够前瞻性地模拟SARS-CoV-2的进化。
有关扩散模型的使用,请参考此代码库:https://github.com/da03/hierarchical_diffusion_LM
高性能计算
我们有一个CLI工具,可以更轻松地在各种HPC平台上启动训练作业。您可以通过指定-T, --template选项来指定要提交到的系统。我们目前有polaris和perlmutter的模板。默认情况下,提交的作业将输出结果到运行提交命令的目录,您可以使用-w选项指定不同的workdir。请运行python -m genslm.hpc.submit --help获取更多信息。有关yaml选项的文档,请参阅config.py,并注意config.yaml路径必须是绝对路径。
module load conda/2022-07-19
conda activate genslm
python -m genslm.hpc.submit -T polaris -a gpu_hack -q debug -t 00:10:00 -n 1 -j test-job-0 -v "-c config.yaml"
模块特定参数通过-v标志逐字传递,参数必须在引号内。
有关其他命令,请参阅COMMANDS.md。
贡献
请通过问题跟踪器报告错误、增强请求或问题。
如果您想贡献,请参阅CONTRIBUTING.md。
许可证
genslm采用MIT许可证,详见LICENSE.md文件。
引用
如果您在研究中使用我们的模型,请引用以下论文:
@article{zvyagin2022genslms,
title={GenSLMs: Genome-scale language models reveal SARS-CoV-2 evolutionary dynamics.},
author={Zvyagin, Max T and Brace, Alexander and Hippe, Kyle and Deng, Yuntian and Zhang, Bin and Bohorquez, Cindy Orozco and Clyde, Austin and Kale, Bharat and Perez-Rivera, Danilo and Ma, Heng and others},
journal={bioRxiv},
year={2022},
publisher={Cold Spring Harbor Laboratory}
}

 访问官网
访问官网 Github
Github 文档
文档 论文
论文










