PaCMAP
目录
简介
我们的工作已发表在机器学习研究期刊(JMLR)上!
PaCMAP(成对控制流形近似)是一种可用于可视化的降维方法,可以同时保留原始空间中数据的局部和全局结构。PaCMAP通过优化三种类型的点对来实现低维嵌入:邻居对(pair_neighbors)、中距离对(pair_MN)和远距离对(pair_FP)。
以往的降维技术要么专注于局部结构(如t-SNE、LargeVis和UMAP),要么专注于全局结构(如TriMAP),但很少同时考虑两者,尽管可以通过仔细调整算法中控制全局和局部结构平衡的参数来实现,这主要是通过调整考虑的邻居数量。PaCMAP不是考虑更多邻居来保留全局结构,而是动态使用一种特殊的点对组 - 中距离对,首先捕捉全局结构,然后细化局部结构,从而同时保留全局和局部结构。有关本工作的详细背景和讨论,请阅读我们的论文。
发布说明
请参阅发布说明。
安装
通过conda或mamba从conda-forge安装
你可以使用conda或mamba从conda-forge通道安装PaCMAP。
conda:
conda install pacmap -c conda-forge
mamba:
mamba install pacmap -c conda-forge
通过pip从PyPI安装
你可以使用pip从PyPI安装pacmap。它会自动为你安装依赖项:
pip install pacmap
如果在安装依赖项时遇到任何问题,例如Failed building wheel for annoy,你可以尝试使用conda或mamba安装这些依赖项。用户还报告在某些情况下,你可能需要使用numba >= 0.57。
conda install -c conda-forge python-annoy
pip install pacmap
使用方法
在Python中使用PaCMAP
pacmap包设计为与scikit-learn兼容,这意味着它具有与sklearn.manifold模块中函数类似的接口。要在你自己的数据集上运行pacmap,你应该按照安装中的说明安装该包,然后导入模块。以下代码片段包括了如何在COIL-20数据集上使用PaCMAP的用例:
import pacmap
import numpy as np
import matplotlib.pyplot as plt
# 加载预处理过的coil_20数据集
# 你可以将其更改为任何ndarray格式的数据集,其形状为(N, D)
# 其中N是样本数,D是每个样本的维度
X = np.load("./data/coil_20.npy", allow_pickle=True)
X = X.reshape(X.shape[0], -1)
y = np.load("./data/coil_20_labels.npy", allow_pickle=True)
# 初始化pacmap实例
# 将n_neighbors设置为"None"会导致自动选择,如下面的"参数"部分所示
embedding = pacmap.PaCMAP(n_components=2, n_neighbors=10, MN_ratio=0.5, FP_ratio=2.0)
# 拟合数据(转换后数据的索引对应原始数据的索引)
X_transformed = embedding.fit_transform(X, init="pca")
# 可视化嵌入结果
fig, ax = plt.subplots(1, 1, figsize=(6, 6))
ax.scatter(X_transformed[:, 0], X_transformed[:, 1], cmap="Spectral", c=y, s=0.6)
在R中使用PaCMAP
你也可以使用reticulate包在R中使用PaCMAP。我们提供了一个示例R notebook,演示了如何在R中调用PaCMAP进行可视化。
性能基准
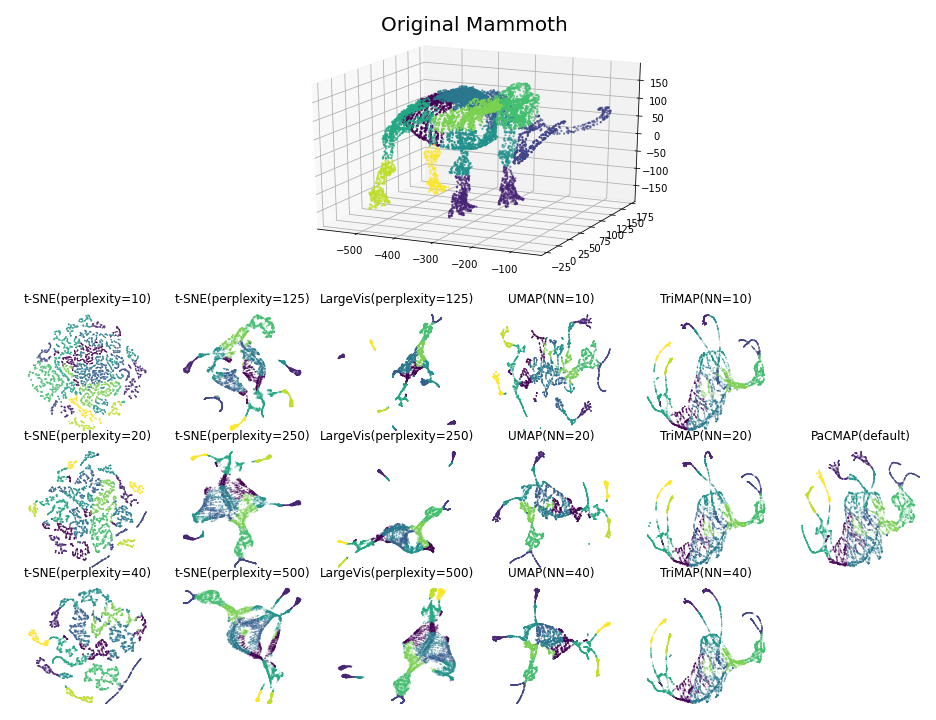
以下图像是两个数据集的可视化结果:MNIST(n=70,000,d=784)和Mammoth(n=10,000,d=3),由PaCMAP生成。这两个可视化分别展示了PaCMAP保留局部和全局结构的能力。

参数
以下列出了最重要的参数。改变这些值会显著影响降维结果,具体说明见我们论文的8.3节。
-
n_components:输出的维度数。默认为2。 -
n_neighbors:k-最近邻图中考虑的邻居数。默认为10。我们还允许将此参数设置为None以启用自动选择邻居数:对于样本数小于10000的数据集,邻居数将设置为10。对于样本数(n)大于10000的大型数据集,该值为:10 + 15 * (log10(n) - 4)。 -
MN_ratio:中距离对数量与邻居数量的比率,n_MN=n_neighbors * MN_ratio。默认为0.5。
-
FP_ratio:远距离对数量与邻居数量的比率,n_FP=n_neighbors * FP_ratio。默认为2。 初始化对结果也很重要,但它是
fit和fit_transform函数的参数。 -
init:低维嵌入的初始化。可选"pca"或"random",或用户提供的形状为 (N, 2) 的 numpy ndarray。默认为"random"。
其他参数包括:
num_iters:迭代次数。默认为 450。对大多数数据集来说,450 次迭代足以收敛。pair_neighbors、pair_MN和pair_FP:预先指定的邻居对、中近点和远点对。允许用户使用自己的图。默认为None。verbose:打印 pacmap 的进度。默认为False。lr:AdaGrad 优化器的学习率。默认为 1。apply_pca:pacmap 在构建 k 近邻图之前是否应对数据应用 PCA。使用 PCA 预处理数据可以在不损失太多精度的情况下大大加快降维过程。注意,此选项不影响优化过程的初始化。intermediate:pacmap 是否也输出低维嵌入优化过程的中间阶段。如果为True,则输出将是大小为 (n,n_components, 13) 的 numpy 数组,其中每个切片是特定步数下输出嵌入的"快照",步数为 [0, 10, 30, 60, 100, 120, 140, 170, 200, 250, 300, 350, 450]。
方法
与 scikit-learn API 类似,PaCMAP 实例可以通过 fit、fit_transform 和 transform 方法为数据集生成嵌入。我们目前支持 numpy.ndarray 格式作为输入。具体来说,要将 pandas DataFrame 转换为 ndarray 格式,请参阅 pandas 文档。有关更详细的演示,请参见 demo 目录。
如何使用用户指定的最近邻
在 0.4 版本中,我们提供了一个新选项,允许用户在映射大规模数据集时使用自己的最近邻。有关如何使用用户指定的最近邻与 PaCMAP 一起使用的详细演示,请参见 demo。
复现我们的实验
我们提供了用于运行实验的代码,以便更好地复现。代码分为三个部分,分别位于三个文件夹中:
data,包含我们使用的所有数据集,预处理成每种降维方法使用的文件格式。注意,由于小鼠单细胞 RNA 序列数据集太大(约 4GB),你可能需要从链接下载。MNIST 和 FMNIST 数据集已压缩,使用前需要解压。COIL-100 数据集即使压缩后仍然太大,请使用 Preprocessing.ipynb 文件自行预处理。experiments,包含我们用于生成降维结果的所有脚本。evaluation,包含我们用于评估降维结果的所有脚本,如论文第 8 节所述。
下载代码后,你可能需要指定脚本中的一些路径以使其完全可用。
引用
如果你在出版物中使用了 PaCMAP,或使用了此存储库中的实现,请使用以下 bibtex 引用我们的论文:
@article{JMLR:v22:20-1061,
author = {Yingfan Wang and Haiyang Huang and Cynthia Rudin and Yaron Shaposhnik},
title = {Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization},
journal = {Journal of Machine Learning Research},
year = {2021},
volume = {22},
number = {201},
pages = {1-73},
url = {http://jmlr.org/papers/v22/20-1061.html}
}
关于 PaCMAP 在生物数据集上的表现,请查看以下论文:
@article{huang2022towards,
title={Towards a comprehensive evaluation of dimension reduction methods for transcriptomic data visualization},
author={Huang, Haiyang and Wang, Yingfan and Rudin, Cynthia and Browne, Edward P},
journal={Communications biology},
volume={5},
number={1},
pages={719},
year={2022},
publisher={Nature Publishing Group UK London}
}
许可证
请参阅许可证文件。

 访问官网
访问官网 Github
Github 文档
文档 论文
论文










