
Meditron是一套开源医疗大型语言模型(LLMs)。
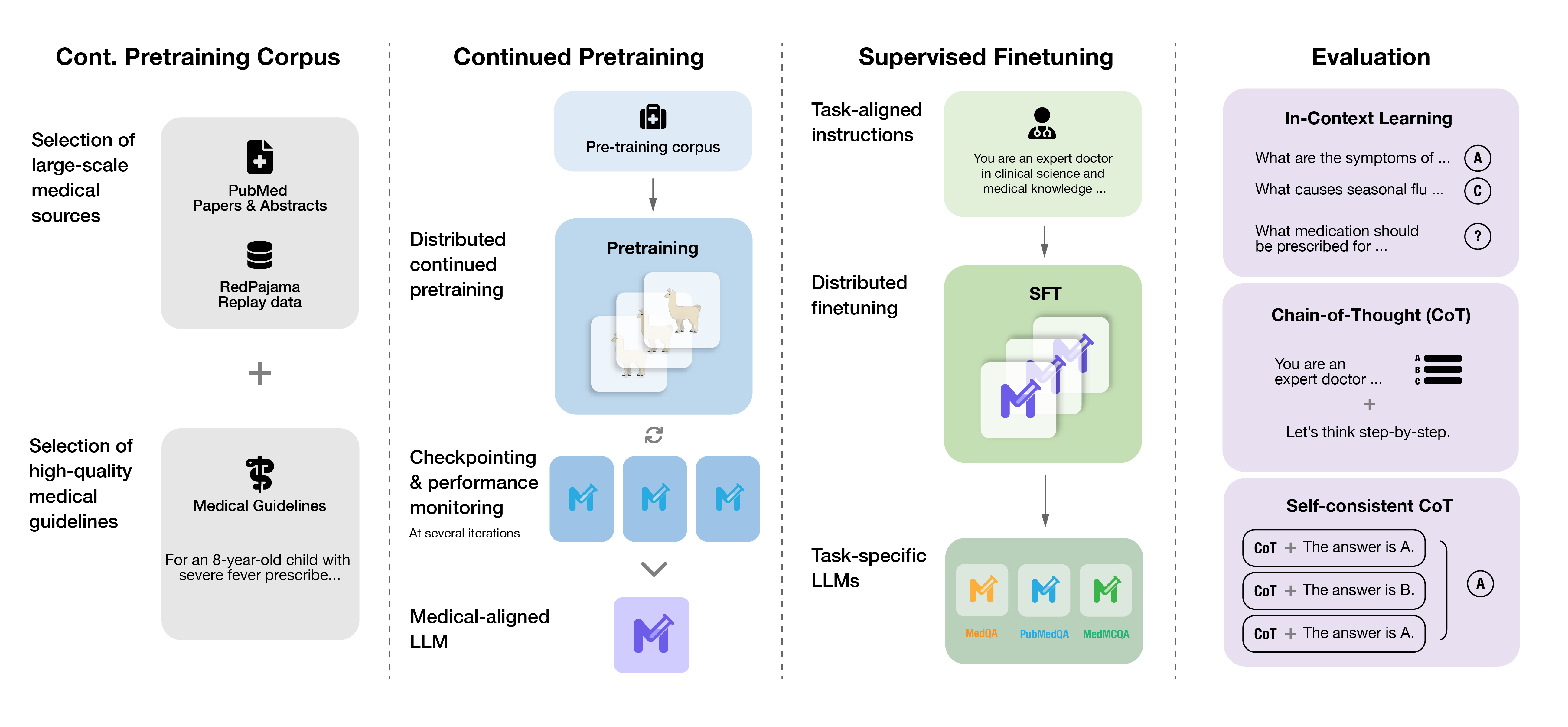
我们发布了Meditron-7B和Meditron-70B,这两个模型是通过在全面策划的医学语料库上继续预训练来从Llama-2适应到医学领域的。该语料库包括精选的PubMed论文和摘要、一个新的国际认可的医疗指南数据集以及一般领域语料库。
经过相关数据微调后,Meditron-70B在多项医学推理任务上的表现优于Llama-2-70B、GPT-3.5和Flan-PaLM。
注意事项
虽然Meditron旨在从高质量证据来源编码医学知识,但它尚未适应于适当、安全或在专业可操作约束内传递这些知识。我们建议在没有进行广泛的用例调整以及额外测试(特别包括在真实世界实践环境中的随机对照试验)的情况下,不要在医疗应用中使用Meditron。
模型详情
- 开发者: EPFL LLM团队
- 模型类型: 因果解码器-仅transformer语言模型
- 语言: 主要为英语
- 模型许可: LLAMA 2社区许可协议
- 代码许可: APACHE 2.0许可
- 继续预训练自模型: Llama-2-70B
- 上下文长度: 4k令牌
- 输入: 仅文本数据
- 输出: 模型仅生成文本
- 状态: 这是一个在离线数据集上训练的静态模型。随着我们提高模型性能,将发布调优模型的未来版本。
- 知识截止日期: 2023年8月
- 训练器: epflLLM/Megatron-LLM
- 论文: Meditron-70B:扩展大型语言模型的医学预训练
如何使用
您可以直接从HuggingFace模型中心加载Meditron模型,如下所示:
from transformers import AutoTokenizer, AutoModelForCausalLM
tokenizer = AutoTokenizer.from_pretrained("epfl-llm/meditron-70b")
model = AutoModelForCausalLM.from_pretrained("epfl-llm/meditron-70b")
医学训练数据
我们发布了用于下载和预处理Meditron训练数据的代码。
MediTron的领域适应性预训练语料库GAP-Replay结合了来自四个语料库的481亿个标记:
- 临床指南:一个新的语料库,包含46K条临床实践指南,来自各种医疗相关来源,包括医院和国际组织,
- 论文摘要:从受限访问的PubMed和PubMed Central论文中提取的1610万篇摘要,
- 医学论文:从500万篇公开可用的PubMed和PubMed Central论文中提取的全文文章。
- 重放数据集:从RedPajama-v1中采样的4亿个标记的通用领域预训练数据。
下载说明
您可以通过在gap-replay文件夹中运行./download.sh来下载和预处理整个GAP-Replay语料库。
您可以从HuggingFace数据集中心下载我们的指南语料库中的36K开放访问文章。
from datasets import load_dataset
dataset = load_dataset("epfl-llm/guidelines")
您可以通过在guidelines文件夹中运行./download.sh来抓取和清理所有46K条指南(包括受限访问来源)。
更多详细信息可以在GAP-Replay文档中找到。
训练程序
我们使用Megatron-LLM分布式训练库(Nvidia的Megatron LM项目的衍生版本)来优化训练效率。 硬件包括16个节点,每个节点有8个NVIDIA A100(80GB)SXM GPU,通过NVLink和NVSwitch连接,配备单个Nvidia ConnectX-6 DX网卡,2个AMD EPYC 7543 32核处理器和512 GB RAM。 节点通过RDMA over Converged Ethernet连接。
我们的三向并行方案使用以下内容:
- 数据并行(DP -- 不同的GPU处理批次的不同子集)为2,
- 流水线并行(PP -- 不同的GPU处理不同的层)为8,
- 张量并行(TP -- 不同的GPU处理矩阵乘法的不同子张量)为8。
训练超参数(7B)
| bf16 | true |
| lr | 3e-4 |
| eps | 1e-5 |
| betas | [0.9, 0.95] |
| clip_grad | 1 |
| weight decay | 0.1 |
| DP size | 16 |
| TP size | 4 |
| PP size | 1 |
| seq length | 2048 |
| lr scheduler | cosine |
| min lr | 1e-6 |
| warmup iteration | 2000 |
| micro batch size | 10 |
| global batch size | 1600 |
训练超参数(70B)
| bf16 | true |
| lr | 1.5e-4 |
| eps | 1e-5 |
| betas | [0.9, 0.95] |
| clip_grad | 1 |
| weight decay | 0.1 |
| DP size | 2 |
| TP size | 8 |
| PP size | 8 |
| seq length | 4096 |
| lr scheduler | cosine |
| min lr | 1e-6 |
| warmup iteration | 2000 |
| micro batch size | 2 |
| global batch size | 512 |
您可以在这里查看我们通过Megatron-LLM预训练模型所使用的脚本:finetune.sh
有监督微调
我们再次使用Megatron-LLM分布式训练库进行有监督微调(单节点和多节点)。
我们创建了一个文件sft.py,它通过Megatron-LLM自动处理标记化和微调过程。要启动多节点微调过程,这里有一个例子:
cd finetuning
python sft.py \
--checkpoint=baseline \
--size=70 \
--run_name=cotmedqa \
--data /pure-mlo-scratch/zechen/meditron/benchmarks/ft_preprocessed/medqa_cot_train.jsonl \
--val /pure-mlo-scratch/zechen/meditron/benchmarks/ft_preprocessed/medqa_cot_validation.jsonl \
--micro_batch=4
--nodes=4 \
--addr=<RANK0_HOST_NAME> \
--save_interval=200 \
--pp=4 \
--seq 4096 \
--rank=<CURRENT_RANK>
在节点rank-0、rank-1、rank-2和rank-3上运行上述代码行,以启动4节点微调过程。
重要提示:确保在sft.py和finetune_sft.sh中定义了正确的路径。
微调超参数
| bf16 | true |
| 学习率 | 2e-5 |
| eps | 1e-5 |
| betas | [0.9, 0.95] |
| 梯度裁剪 | 1 |
| 权重衰减 | 0.1 |
| 数据并行大小 | 16 |
| 张量并行大小 | 4 |
| 流水线并行大小 | 1 |
| 序列长度 | 2048 或 4096 |
| 学习率调度器 | 余弦 |
| 最小学习率 | 2e-6 |
| 预热比例 | 0.1 |
| 添加的标记 | [< |
用途
Meditron-70B 正在进行进一步测试和评估,作为一个人工智能助手,以增强临床决策并使医疗保健领域的大型语言模型更加普及。潜在的用例可能包括但不限于:
- 医学考试问题回答
- 支持鉴别诊断
- 疾病信息(症状、原因、治疗)查询
- 一般健康信息查询
可以使用此模型生成文本,这对于实验和了解其功能很有用。不应直接将其用于可能影响他人的生产或工作。
我们不建议在生产环境中使用此模型进行自然语言生成,无论是否经过微调。
下游使用
Meditron-70B 和 Meditron-7B 都是未经微调或指令调整的基础模型。它们可以针对特定的下游任务和应用进行微调、指令调整或 RLHF 调整。 我们有两种方法使用此模型进行下游问答任务:
- 我们在提示中应用上下文学习,添加 k 个示例(在我们的论文中为 3 或 5 个)。
- 我们使用特定的训练集对模型进行微调,以适应下游问答任务。
我们鼓励并期待基础模型能被应用于更多样化的应用。
如果您想以更互动的方式提示模型,我们建议使用具有支持聊天和文本生成的用户界面的高吞吐量、内存效率高的推理引擎。
您可以查看我们下面的部署指南,其中我们使用了 FastChat 和 vLLM。我们通过交互式用户界面平台 BetterChatGPT 收集了用于定性分析的生成内容。以下是我们使用的提示格式示例:

医学基准推理与评估
要求
在开始之前,请安装必要的包:
vllm >= 0.2.1
transformers >= 4.34.0
datasets >= 2.14.6
torch >= 2.0.1
有关使用医学基准进行推理和评估的详细说明,请阅读此处的文档 推理和评估说明。
模型部署
有关部署 meditron 模型并进行交互式聊天会话的详细说明,请阅读此处的文档 模型部署
引用
如果您使用本软件或我们的论文,请引用它们:
@misc{chen2023meditron70b,
title={MEDITRON-70B: Scaling Medical Pretraining for Large Language Models},
author={Zeming Chen and Alejandro Hernández-Cano and Angelika Romanou and Antoine Bonnet and Kyle Matoba and Francesco Salvi and Matteo Pagliardini and Simin Fan and Andreas Köpf and Amirkeivan Mohtashami and Alexandre Sallinen and Alireza Sakhaeirad and Vinitra Swamy and Igor Krawczuk and Deniz Bayazit and Axel Marmet and Syrielle Montariol and Mary-Anne Hartley and Martin Jaggi and Antoine Bosselut},
year={2023},
eprint={2311.16079},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
@software{epfmedtrn,
author = {Zeming Chen and Alejandro Hernández-Cano and Angelika Romanou and Antoine Bonnet and Kyle Matoba and Francesco Salvi and Matteo Pagliardini and Simin Fan and Andreas Köpf and Amirkeivan Mohtashami and Alexandre Sallinen and Alireza Sakhaeirad and Vinitra Swamy and Igor Krawczuk and Deniz Bayazit and Axel Marmet and Syrielle Montariol and Mary-Anne Hartley and Martin Jaggi and Antoine Bosselut},
title = {MediTron-70B: Scaling Medical Pretraining for Large Language Models},
month = November,
year = 2023,
url = {https://github.com/epfLLM/meditron}
}

 Github
Github Huggingface
Huggingface 论文
论文










