gos 🦆
gos是一个用于Python的声明式基因组可视化库。它基于Gosling JSON规范构建,为创作交互式基因组可视化提供了简化的接口。
安装
gos API正在积极开发中。欢迎并感谢您的反馈。
pip install gosling[all]
文档
更多信息请参见文档网站。
示例
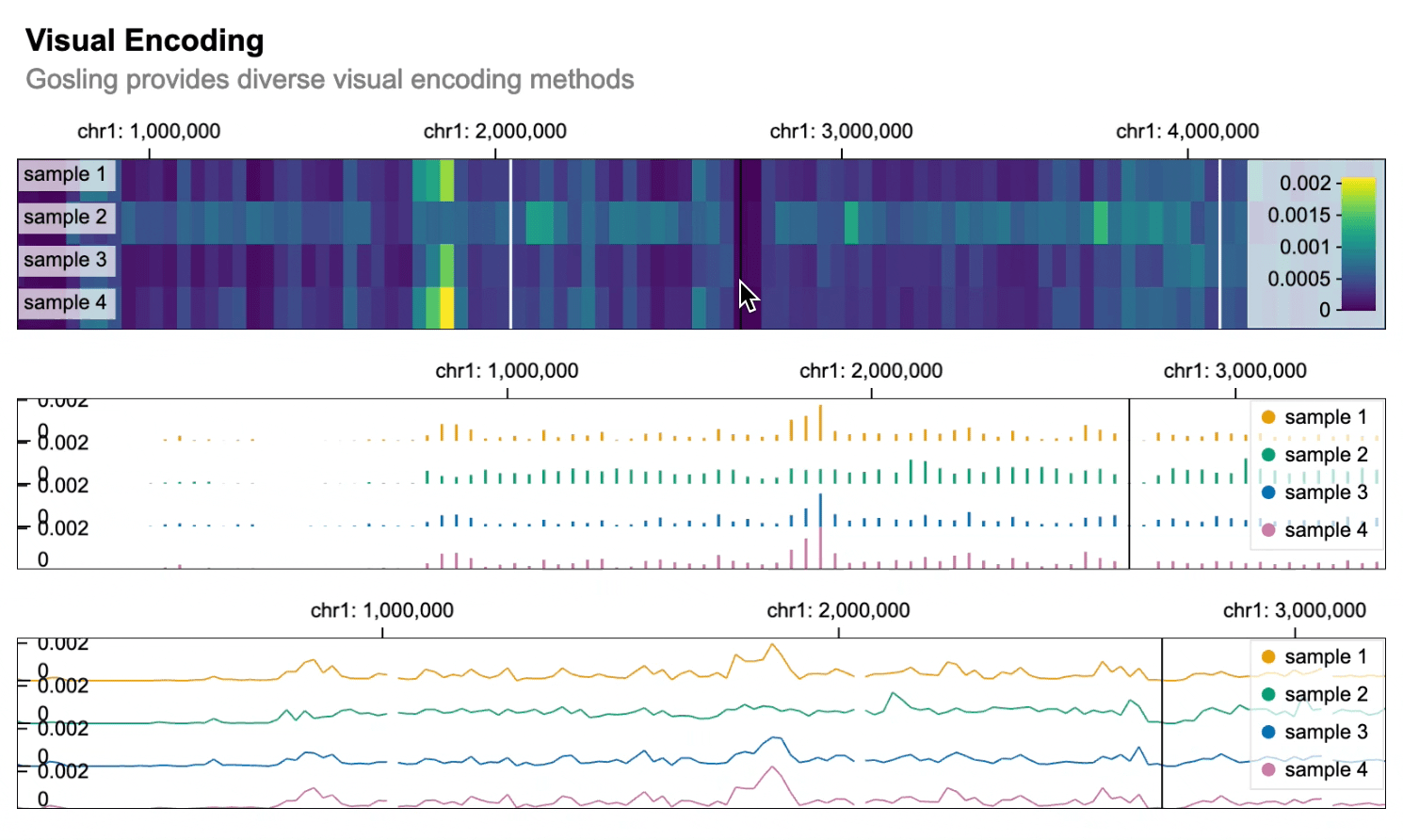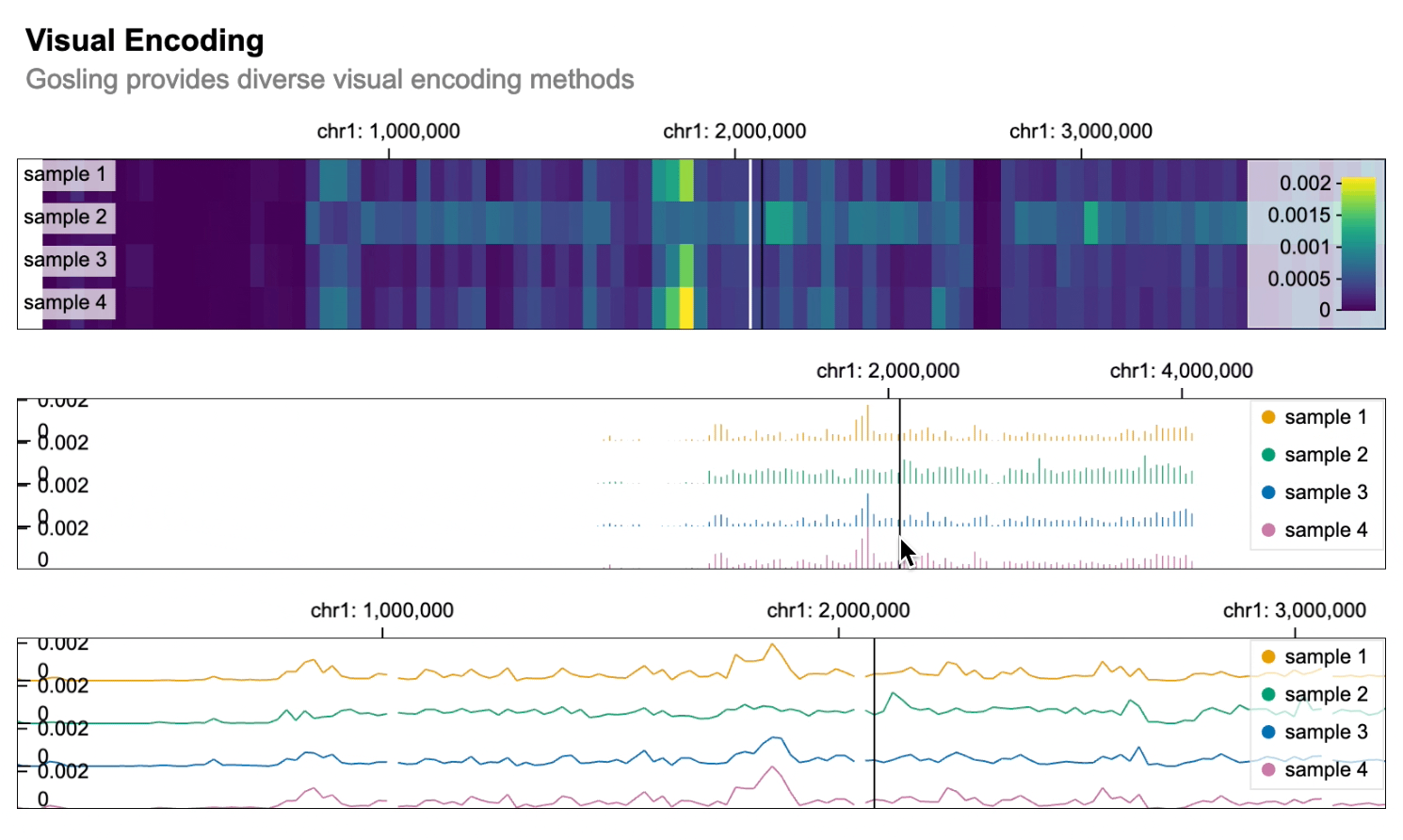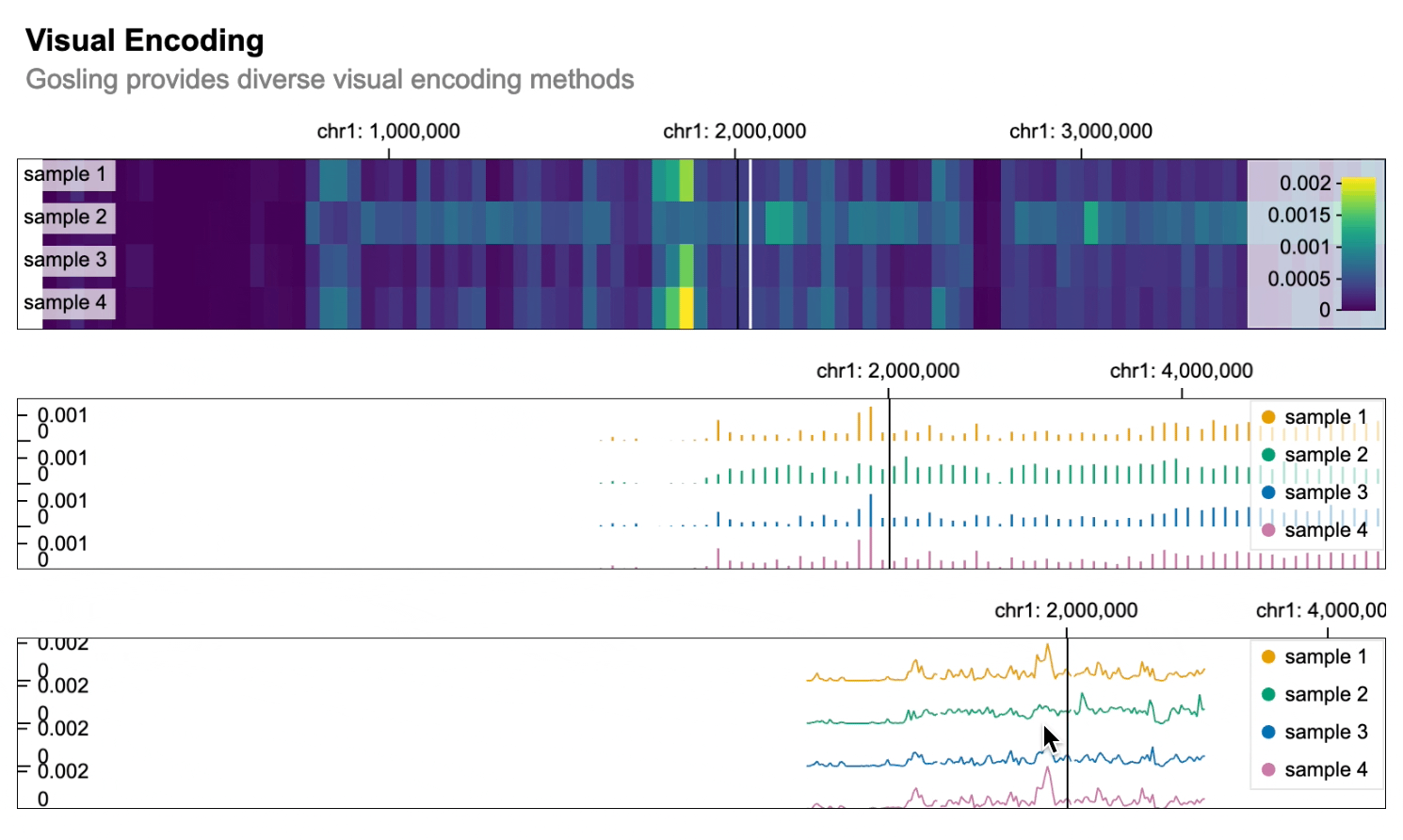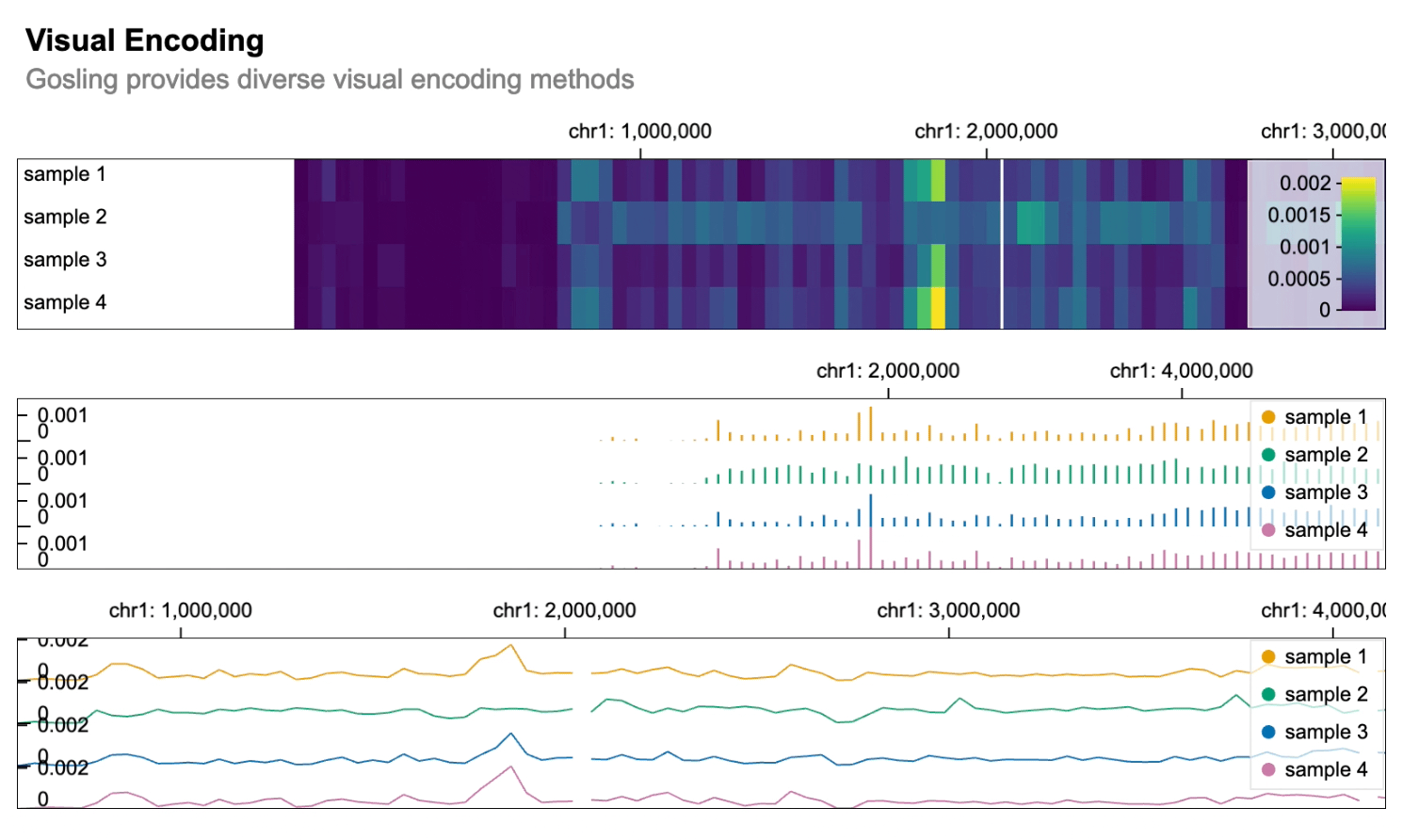
import gosling as gos
data = gos.multivec(
url="https://server.gosling-lang.org/api/v1/tileset_info/?d=cistrome-multivec",
row="sample",
column="position",
value="peak",
categories=["sample 1", "sample 2", "sample 3", "sample 4"],
binSize=5,
)
base_track = gos.Track(data, width=800, height=100)
heatmap = base_track.mark_rect().encode(
x=gos.X("start:G", axis="top"),
xe="end:G",
row=gos.Row("sample:N", legend=True),
color=gos.Color("peak:Q", legend=True),
)
bars = base_track.mark_bar().encode(
x=gos.X("position:G", axis="top"),
y="peak:Q",
row="sample:N",
color=gos.Color("sample:N", legend=True),
)
lines = base_track.mark_line().encode(
x=gos.X("position:G", axis="top"),
y="peak:Q",
row="sample:N",
color=gos.Color("sample:N", legend=True),
)
gos.vertical(heatmap, bars, lines).properties(
title="视觉编码",
subtitle="Gosling提供多种视觉编码方法",
layout="linear",
centerRadius=0.8,
xDomain=gos.GenomicDomain(chromosome="1", interval=[1, 3000500]),
)
示例库
我们已经在gosling/examples/中开始建立一个社区示例库。如果您有兴趣贡献,欢迎提交PR!如果您在寻找灵感,可以查看现有的JSON示例。
开发
pip install -e '.[dev]'
模式绑定(gosling/schema/)和文档(doc/user_guide/API.rst)是自动生成的。请不要直接编辑这些文件。
# 生成gosling/schema/*
python tools/generate_schema_wrapper.py <tag_name>
发布
git checkout main && git pull
git commit -m "v0.[minor].[patch]"
git tag -a v0.[minor].[patch] -m "v0.[minor].[patch]"
git push --follow-tags
设计与实现
gos在项目理念和实现上深受Altair的启发和借鉴。内部Python API是使用直接从Altair改编的代码从Gosling规范自动生成的,用于生成Vega-Lite绑定。这种设计选择保证了可视化与Gosling规范完全一致的类型检查,并且Python API随着时间的推移与不断发展的模式保持一致。特别感谢Jake Vanderplas和其他人在schemapi上的工作。

 访问官网
访问官网 Github
Github 文档
文档










