
粒子生命模拟
这是一个简单的程序,用于模拟原始的人工生命。通过类似原子的粒子之间简单的吸引或排斥规则,产生复杂的自组织类生命模式。除去图形界面元素,代码不到一页。视频教程和详细说明请见下方。
了解更多(YouTube 视频教程):
在线演示(JavaScript 版本):
点击这里查看实时演示(JavaScript):
- 2d - https://hunar4321.github.io/particle-life/particle_life.html
- 3d - https://hunar4321.github.io/particle-life/particle_life_3d.html
界面(C++ 版本)

示例结果

一些有趣的模式复现:
复现这些模式时,参数不需要完全精确。获得有趣模式的最佳方法是先尝试随机参数探索,一旦发现有趣的模式,就尝试逐步微调。为避免陷入局部最优,可以偶尔进行大幅度的参数跳跃。通过这种方式,有趣且不同的模式将不断涌现。
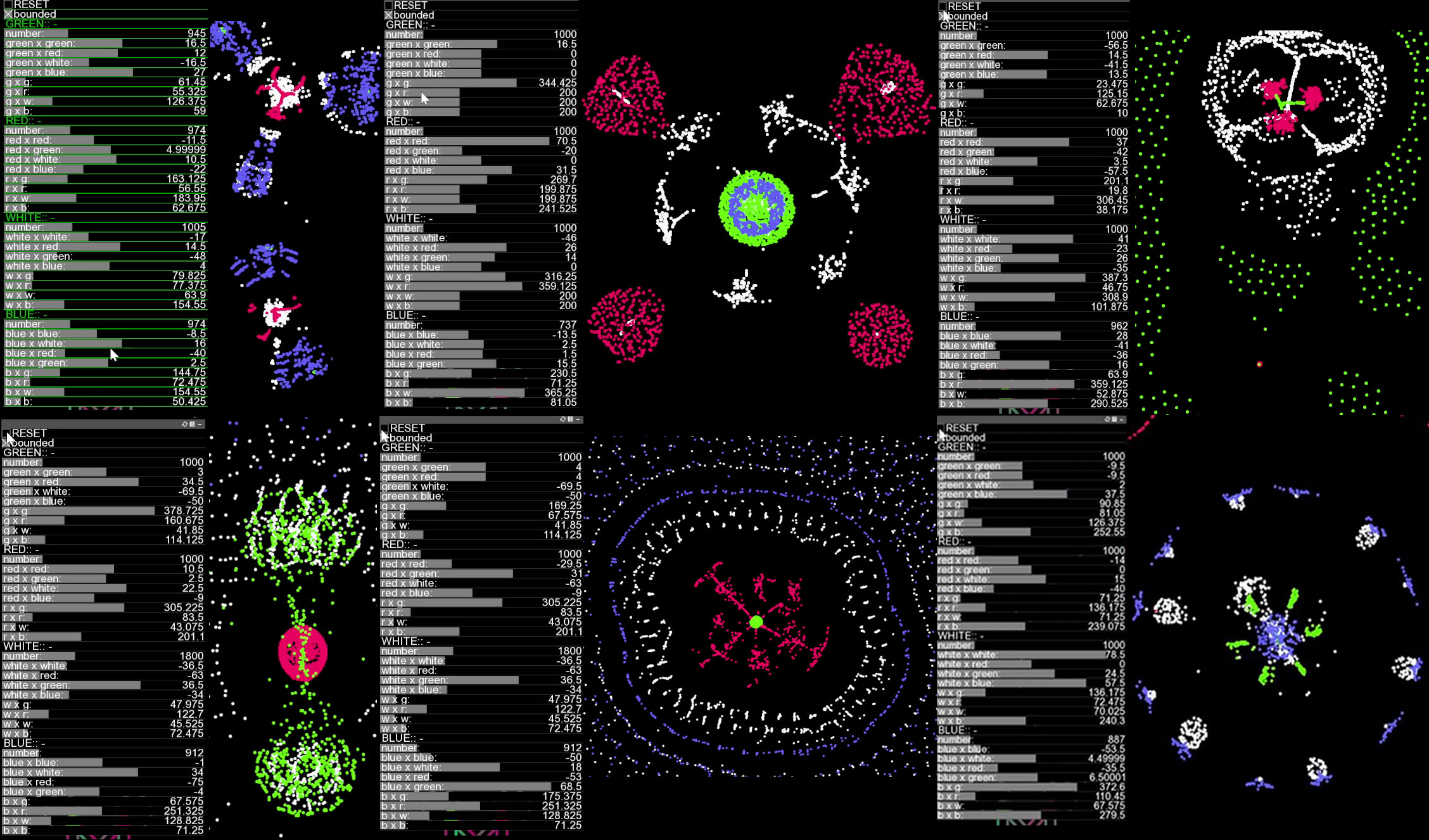
使用方法:
下载此仓库。解压文件后,进入 /particle_life/bin/ 文件夹,点击 particle_life.exe
代码:
源代码有 C++、JavaScript 和 Python 版本。 观看此 YouTube 视频获取详细教程:https://youtu.be/0Kx4Y9TVMGg
如果你想为 C++ 程序做出贡献,核心算法位于 "/particle_life/src/ofApp.cpp" 文件的前 100 行。其余部分是由 openFrameworks 库提供的图形界面组件和渲染控制,这是一个开源且易用的图像渲染库。
首先,下载此仓库,然后从这里下载 openFrameworks 库:https://openframeworks.cc/。使用 openFramework 的 projectGenerator 并将 /particle_life/ 文件夹导入到项目中。
或者,生成一个新的 openFramework 项目并添加 ofxGui。一旦项目文件生成,用这里提供的 /src/ 文件夹替换原有的文件夹。
现在你可以在你的机器上编译 C++ 代码了。
其他移植版本:
JavaScript 代码就像这样简单:
另外,查看 particle_life.html 文件以获取更优化的版本 - 感谢那些做出贡献的人。
<canvas id="life" width="500" height="500"></canvas>
<script>
//Hunar Ahmad @ brainxyz
m = document.getElementById("life").getContext("2d");
draw = (x, y, c, s) => {
m.fillStyle = c;
m.fillRect(x, y, s, s);
};
atoms = [];
atom = (x, y, c) => {
return { x: x, y: y, vx: 0, vy: 0, color: c };
};
random = () => {
return Math.random() * 400 + 50;
};
create = (number, color) => {
group = [];
for (let i = 0; i < number; i++) {
group.push(atom(random(), random(), color));
atoms.push(group[i]);
}
return group;
};
rule = (atoms1, atoms2, g) => {
for (let i = 0; i < atoms1.length; i++) {
fx = 0;
fy = 0;
for (let j = 0; j < atoms2.length; j++) {
a = atoms1[i];
b = atoms2[j];
dx = a.x - b.x;
dy = a.y - b.y;
d = Math.sqrt(dx * dx + dy * dy);
if (d > 0 && d < 80) {
F = (g * 1) / d;
fx += F * dx;
fy += F * dy;
}
}
a.vx = (a.vx + fx) * 0.5;
a.vy = (a.vy + fy) * 0.5;
a.x += a.vx;
a.y += a.vy;
if (a.x <= 0 || a.x >= 500) { a.vx *= -1; }
if (a.y <= 0 || a.y >= 500) { a.vy *= -1; }
}
};
yellow = create(200, "yellow");
red = create(200, "red");
green = create(200, "green");
update = () => {
rule(green, green, -0.32);
rule(green, red, -0.17);
rule(green, yellow, 0.34);
rule(red, red, -0.1);
rule(red, green, -0.34);
rule(yellow, yellow, 0.15);
rule(yellow, green, -0.2);
m.clearRect(0, 0, 500, 500);
draw(0, 0, "black", 500);
for (i = 0; i < atoms.length; i++) {
draw(atoms[i].x, atoms[i].y, atoms[i].color, 5);
}
requestAnimationFrame(update);
};
update();
</script>
相关主题: 粒子生命模拟、 原始汤 - 进化、 康威生命游戏、 细胞自动机、 自组织模式、
这个项目的灵感来自Jeffery Ventrella的Clusters http://www.ventrella.com/Clusters/。我无法访问Ventrella的代码,但我猜这个项目与其他粒子生命项目的主要区别在于我没有实现碰撞检测,这使得实时模拟数千个粒子成为可能。此外,我添加了GUI控件来实时更改参数,从而可以轻松微调和探索,因此,我能够发现一些从未见过的模式从一些极其简单的关系模型中涌现出来。 这里的代码可能比其他人工生命代码简单一个数量级,因为我最初编写这段代码仅仅是作为非程序员和普通观众的教育材料,以证明复杂性可以从简单性中产生。
待办事项:
- 添加保存和加载参数的功能(这样人们就可以轻松分享他们发现的有趣模型)
- 能够添加更多粒子类型(目前固定为四种粒子类型)
- 目前,最大的瓶颈是嵌套的for循环(计算所有粒子之间的成对距离),使计算复杂度呈二次方增长。如果我们能找到解决方法就太棒了。
- 作为第3点的替代方案,计算成对距离是极易并行的,因此可以在GPU上计算。
- 添加调整屏幕大小的功能,并改进边界检查,因为许多快速移动的粒子可能会逃离屏幕边界。
- 添加更直观的UI,以便对参数进行更精细的控制。
- 添加一个随机化按钮,或者更好的是,有一个简单的元规则来持续和递归地改变初始规则。这样,模式就永远不会陷入局部最大值,并且会不断变化!
- 更好的微调方法是使用进化算法来选择和优化参数,但需要为此编写一个适应度函数。我目前不知道适应度函数在这个程序的领域中对应什么。在我们的世界中,适应度函数是竞争和适者生存。然而,在这里,我们微调和选择产生有趣模式的参数,但"有趣"这个词说起来容易,定义起来难!

 访问官网
访问官网 Github
Github











