欢迎使用全新的nnU-Net!
如果您想查看旧版本,请点击这里。
从V1版本迁移过来?请查看简明迁移指南。强烈建议您仍然阅读其余文档 ;-)
2024-04-18 更新:新的残差编码器UNet预设可用!
残差编码器UNet预设大幅提高了分割性能。 它们适用于各种GPU内存目标。保证都是很棒的功能! 点击这里了解更多详情。
同时也请查看我们关于系统地对医学图像分割领域最新发展进行基准测试的新论文。结果可能会让您感到惊讶!
nnU-Net是什么?
图像数据集具有极大的多样性:图像维度(2D、3D)、模态/输入通道(RGB图像、CT、MRI、显微镜等)、图像大小、体素大小、类别比例、目标结构属性等在不同数据集之间存在显著差异。传统上,面对新问题时需要手动设计和优化定制解决方案 - 这个过程容易出错、不可扩展,成功与否在很大程度上取决于实验者的技能。即便对专家而言,这个过程也绝非易事:不仅需要考虑许多设计选择和数据属性,而且它们之间还紧密相连,使得可靠的手动流程优化几乎不可能实现!
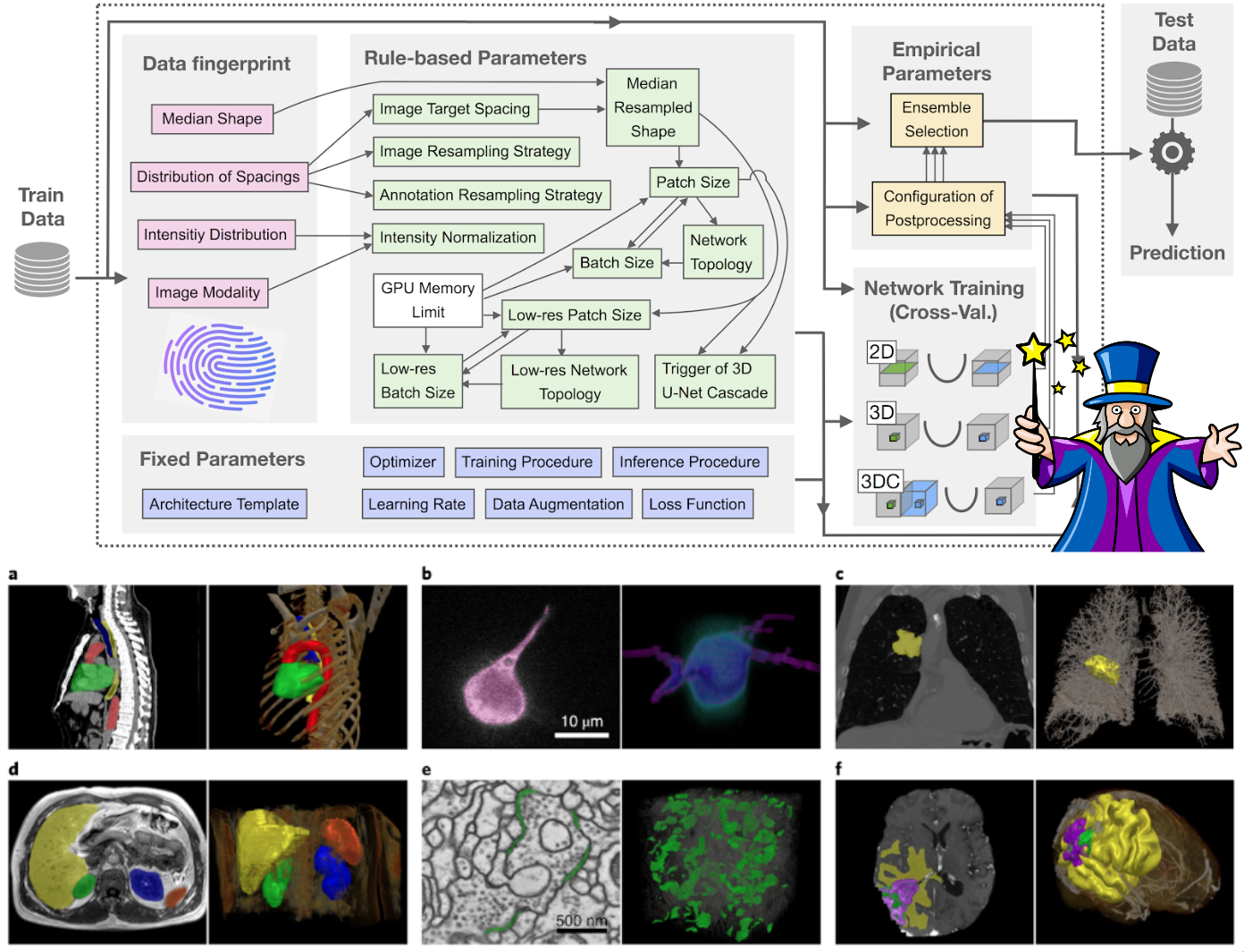
nnU-Net是一种能够自动适应给定数据集的语义分割方法。它会分析提供的训练样例,并自动配置匹配的基于U-Net的分割流程。您无需任何专业知识!您只需训练模型并将其用于您的应用。
发布时,nnU-Net在生物医学领域的23个竞赛数据集上进行了评估。尽管与每个数据集的手工定制解决方案竞争,nnU-Net的全自动流程仍在多个公开排行榜上获得第一名!自那以后,nnU-Net经受住了时间的考验:它继续被用作基线和方法开发框架(2020年MICCAI 10个挑战中有9个获胜者和2021年MICCAI 7个挑战中有5个获胜者基于nnU-Net构建他们的方法,我们用nnU-Net赢得了AMOS2022)!
使用nnU-Net时,请引用以下论文:
Isensee, F., Jaeger, P. F., Kohl, S. A., Petersen, J., & Maier-Hein, K. H. (2021). nnU-Net: a self-configuring
method for deep learning-based biomedical image segmentation. Nature methods, 18(2), 203-211.
nnU-Net能为您做什么?
如果您是寻求分析自己图像的领域科学家(生物学家、放射科医生等),nnU-Net提供了一个开箱即用的解决方案,几乎可以保证在您的特定数据集上提供出色的结果。只需将您的数据集转换为nnU-Net格式,即可享受AI的强大功能 - 无需任何专业知识!
如果您是开发分割方法的AI研究人员,nnU-Net:
- 提供了一个出色的开箱即用的基线算法,可作为竞争对手
- 可以作为方法开发框架,让您在大量数据集上测试您的贡献,而无需调整单独的流程(例如评估新的损失函数)
- 为进一步的数据集特定优化提供了一个强大的起点。这在参与分割挑战时特别有用
- 为分割方法的设计提供了新的视角:也许您可以找到数据集属性与最佳分割流程之间更好的联系?
nnU-Net的适用范围是什么?
nnU-Net专为语义分割而设计。它可以处理具有任意输入模态/通道的2D和3D图像。它能理解体素间距、各向异性,即使在类别高度不平衡的情况下也能保持稳健。
nnU-Net依赖于监督学习,这意味着您需要为您的应用提供训练样例。所需的训练样例数量因分割问题的复杂程度而异。这里无法给出一个适用于所有情况的数字!由于我们广泛使用数据增强,nnU-Net所需的训练样例可能不比其他解决方案多 - 甚至可能更少。
nnU-Net期望能够在预处理和后处理期间一次性处理整个图像,因此无法处理巨大的图像。作为参考:我们测试了从40x40x40像素到1500x1500x1500的3D图像,以及从40x40到约30000x30000的2D图像!如果您的RAM允许,更大的尺寸也是可能的。
nnU-Net如何工作?
给定一个新数据集,nnU-Net将系统地分析提供的训练样例并创建一个"数据集指纹"。 然后nnU-Net为每个数据集创建几种U-Net配置:
2d:2D U-Net(适用于2D和3D数据集)3d_fullres:在高图像分辨率下运行的3D U-Net(仅适用于3D数据集)3d_lowres→3d_cascade_fullres:3D U-Net级联,其中第一个3D U-Net在低分辨率图像上运行,然后第二个高分辨率3D U-Net细化前者的预测(仅适用于具有大图像尺寸的3D数据集)
请注意,并非所有U-Net配置都会为所有数据集创建。在图像尺寸较小的数据集中,会省略U-Net级联(以及3d_lowres配置),因为全分辨率U-Net的补丁大小已经覆盖了输入图像的很大一部分。
nnU-Net基于三步配方配置其分割流程:
- 固定参数不会被调整。在开发nnU-Net的过程中,我们确定了一个可以始终使用的稳健配置(即某些架构和训练属性)。这包括,例如,nnU-Net的损失函数、(大部分)数据增强策略和学习率。
- 基于规则的参数使用数据集指纹,通过遵循硬编码的启发式规则来调整某些分割流程属性。例如,网络拓扑(池化行为和网络架构深度)会根据补丁大小进行调整;补丁大小、网络拓扑和批量大小会在给定GPU内存约束的情况下联合优化。
- 经验参数本质上是试错。例如,为给定数据集选择最佳的U-Net配置(2D、3D全分辨率、3D低分辨率、3D级联)以及优化后处理策略。
如何开始?
请阅读以下内容:
- 安装说明
- 数据集转换
- 使用说明 附加信息:
- [从稀疏标注中学习(涂鸦、切片)]
- [基于区域的训练]
- [手动数据分割]
- [预训练和微调]
- [nnU-Net中的强度归一化]
- [手动编辑nnU-Net配置]
- [扩展nnU-Net]
- [V2有什么不同?]
竞赛:
- [AutoPET II]
nnU-Net在哪些方面表现良好,哪些方面表现不佳?
nnU-Net在需要从头开始训练的分割问题上表现出色,例如:涉及非标准图像模态和输入通道的研究应用、生物医学领域的挑战数据集、大多数3D分割问题等。我们还没有发现nnU-Net的工作原理失效的数据集!
注意:对于标准分割问题,如ADE20k和Cityscapes中的2D RGB图像,微调基础模型(在大量类似图像上预训练,如Imagenet 22k、JFT-300M)会比nnU-Net提供更好的性能!这只是因为这些模型允许更好的初始化。nnU-Net不支持基础模型,因为1)它们对偏离标准设置的分割问题没有用(见上述数据集),2)通常只支持2D架构,3)与我们为每个数据集仔细调整网络拓扑的核心设计原则相冲突(如果改变拓扑,就无法再转移预训练权重!)
旧版nnU-Net怎么了?
旧版nnU-Net的核心是在2018年参加医学分割十项全能挑战赛期间短时间内拼凑而成的。因此,代码结构和质量并不是最好的。许多功能后来添加,并不完全符合nnU-Net的设计原则。总的来说相当混乱,真的很难用。
nnU-Net V2是一次彻底的改造。是"删除一切重新开始"那种。所以一切都变得更好了(作者认为,哈哈)。虽然分割性能保持不变,但增加了很多很酷的功能。现在也更容易将其用作开发框架,并手动微调其配置以适应新的数据集。重新实现的一个重要驱动因素也是Helmholtz Imaging的出现,促使我们将nnU-Net扩展到更多图像格式和领域。看看这里的一些亮点。
致谢
nnU-Net由Helmholtz Imaging的应用计算机视觉实验室(ACVL)和德国癌症研究中心(DKFZ)的医学图像计算部门开发和维护。

 Github
Github 文档
文档 论文
论文










