UNI
面向计算病理学的通用基础模型
自然医学 
摘要: 组织图像的定量评估对计算病理学(CPath)任务至关重要,需要从全幻灯片图像(WSIs)中客观地描述组织病理学实体。WSIs的高分辨率和形态特征的可变性带来了重大挑战,使得大规模数据标注变得复杂,难以应用于高性能应用。为应对这一挑战,目前的努力提出通过从自然图像数据集迁移学习或在公开可用的组织病理学数据集上进行自监督学习来使用预训练的图像编码器,但尚未在不同组织类型上进行大规模的广泛开发和评估。我们推出了UNI,一个用于病理学的通用自监督模型,使用来自20种主要组织类型的超过100,000张诊断性H&E染色WSIs(>77 TB数据)中的1亿多张图像进行预训练。该模型在34个具有不同诊断难度的代表性CPath任务上进行了评估。除了超越以往最先进的模型外,我们还展示了CPath中的新建模能力,如与分辨率无关的组织分类、使用少样本类原型进行幻灯片分类,以及在OncoTree分类系统中分类多达108种癌症类型的疾病亚型泛化能力。UNI在CPath中推进了大规模无监督表示学习,无论是在预训练数据还是下游评估方面,能够实现数据高效的人工智能模型,可以泛化并迁移到解剖病理学中广泛的具有诊断挑战性的任务和临床工作流程。
什么是UNI?
UNI是为组织病理学开发的最大预训练视觉编码器(1亿张图像,10万张WSIs),在内部肿瘤、感染、炎症和正常组织上开发并公开可用。我们在34个临床任务中展示了最先进的性能,在罕见和代表性不足的癌症类型上表现出显著的性能提升。
- 为什么使用UNI?:UNI不使用开放数据集和大型公共组织学幻灯片集合(TCGA、CPTAC、PAIP、CAMELYON、PANDA和TCIA中的其他数据集)进行预训练,这些数据集通常用于计算病理学中的基准开发。我们向研究界提供UNI,用于构建和评估病理AI模型,而不会在公共基准或私人组织病理学幻灯片集合上造成数据污染风险。
更新
- 2024年7月16日:包含与Virchow的比较。
- 2024年6月15日:包含与Prov-GigaPath的比较。
安装
首先克隆仓库并进入目录:
git clone https://github.com/mahmoodlab/UNI.git
cd UNI
然后创建一个conda环境并安装依赖项:
conda create -n UNI python=3.10 -y
conda activate UNI
pip install --upgrade pip # 启用PEP 660支持
pip install -e .
1. 获取访问权限
从Huggingface模型页面请求访问模型权重:https://huggingface.co/mahmoodlab/UNI。
2. 下载权重 + 创建模型
通过身份验证(使用huggingface_hub)后,可以使用timm库直接加载UNI的ViT-L/16模型架构、预训练权重和图像变换。这种方法会自动将模型权重下载到您主目录中的huggingface_hub缓存(~/.cache/huggingface/hub/models--MahmoodLab--UNI),使用以下命令时timm会自动找到:
import timm
from timm.data import resolve_data_config
from timm.data.transforms_factory import create_transform
from huggingface_hub import login
login() # 使用您的用户访问令牌登录,可在https://huggingface.co/settings/tokens找到
# 需要pretrained=True来加载UNI权重(并首次下载权重)
# 需要传入init_values以成功加载LayerScale参数(例如 - block.0.ls1.gamma)
model = timm.create_model("hf-hub:MahmoodLab/uni", pretrained=True, init_values=1e-5, dynamic_img_size=True)
transform = create_transform(**resolve_data_config(model.pretrained_cfg, model=model))
model.eval()
您也可以将模型权重下载到本地目录中指定的检查点位置。timm库仍用于定义ViT-L/16模型架构。需要手动加载和定义UNI的预训练权重和图像变换。
import os
import torch
from torchvision import transforms
import timm
from huggingface_hub import login, hf_hub_download
login() # 使用您的用户访问令牌登录,可在https://huggingface.co/settings/tokens找到
local_dir = "../assets/ckpts/vit_large_patch16_224.dinov2.uni_mass100k/"
os.makedirs(local_dir, exist_ok=True) # 如果目录不存在则创建
hf_hub_download("MahmoodLab/UNI", filename="pytorch_model.bin", local_dir=local_dir, force_download=True)
model = timm.create_model(
"vit_large_patch16_224", img_size=224, patch_size=16, init_values=1e-5, num_classes=0, dynamic_img_size=True
)
model.load_state_dict(torch.load(os.path.join(local_dir, "pytorch_model.bin"), map_location="cpu"), strict=True)
transform = transforms.Compose(
[
transforms.Resize(224),
transforms.ToTensor(),
transforms.Normalize(mean=(0.485, 0.456, 0.406), std=(0.229, 0.224, 0.225)),
]
)
model.eval()
函数get_encoder执行上述命令,将检查点下载到此GitHub仓库的./assets/ckpts/相对路径中。
from uni import get_encoder
model, transform = get_encoder(enc_name='uni', device=device)
3. 运行推理
您可以使用UNI预训练编码器从组织病理学ROI提取特征,如下所示:
from PIL import Image
image = Image.open("uni.jpg")
image = transform(image).unsqueeze(dim=0) # 图像(torch.Tensor)的形状为[1, 3, 224, 224],经过调整大小和归一化处理(ImageNet参数)
with torch.inference_mode():
feature_emb = model(image) # 提取的特征(torch.Tensor)形状为[1,1024]
这些预提取的特征随后可用于ROI分类(通过线性探测)、幻灯片分类(通过多实例学习)以及其他机器学习设置。
具体用法概述
我们提供了用于加载模型和进行推理的高级函数。对于模型加载,函数get_encoder执行上述步骤2中的命令,将检查点下载到此GitHub仓库的./assets/ckpts/相对路径中。
from uni import get_encoder
model, transform = get_encoder(enc_name='uni', device=device)
对于推理:
from uni.downstream.extract_patch_features import extract_patch_features_from_dataloader
from uni.downstream.eval_patch_features.linear_probe import eval_linear_probe
from uni.downstream.eval_patch_features.fewshot import eval_knn, eval_fewshot
from uni.downstream.eval_patch_features.protonet import ProtoNet, prototype_topk_vote
有关详细示例,请参阅下面的笔记本。
加载/使用模型的更详细入门代码:
请参阅./notebooks/uni_walkthrough.ipynb,以开始加载和使用模型创建嵌入,以及提取ROI特征和执行ROI分类/检索的示例代码。
比较和额外基准测试
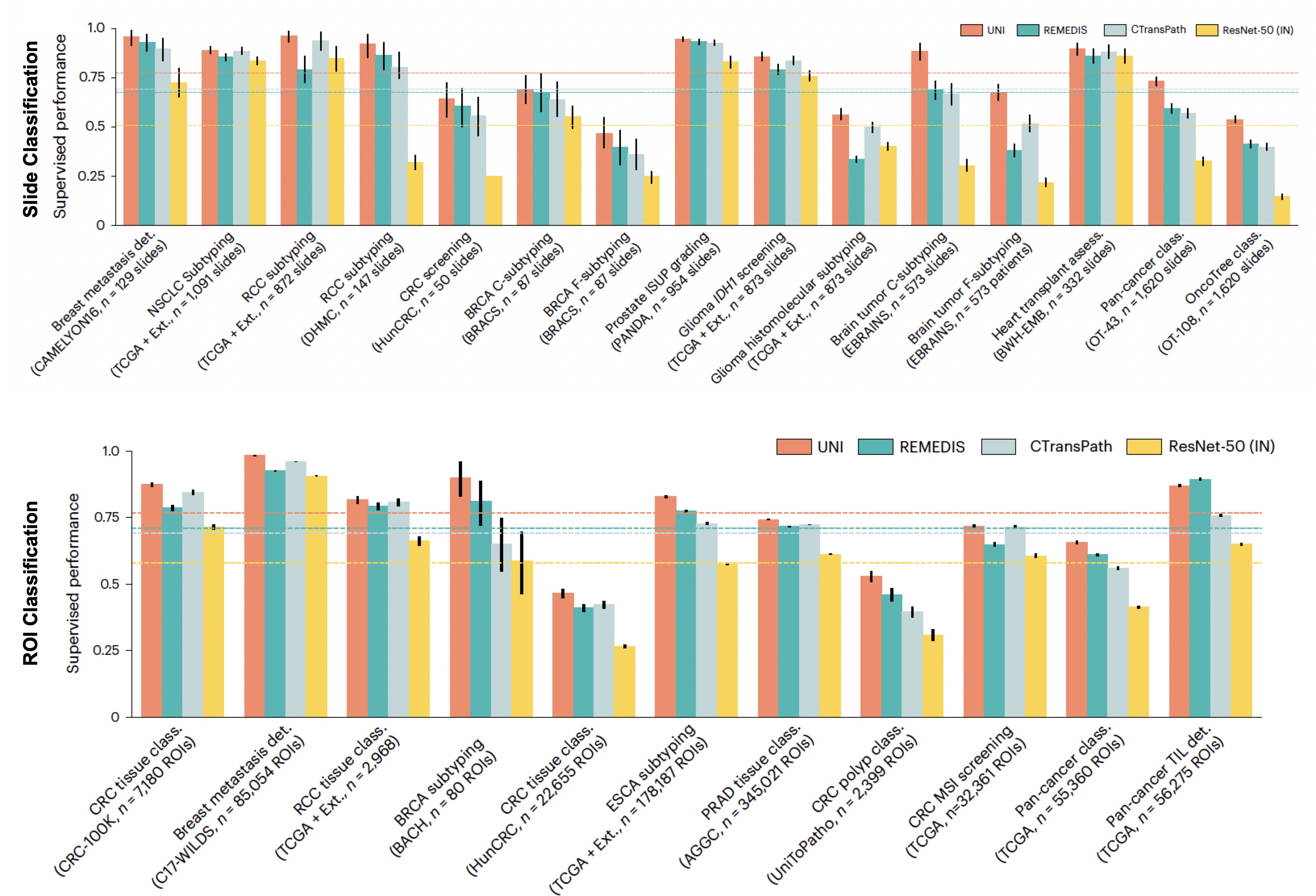
论文[1]中提供了详细的基准测试(如上所示)。一些模型在我们的研究审核期间发布。为了更全面的比较,我们在EBRAINS、PANDA、OncoTree、IHC ER / PR评估、CRC-100K-Raw和TCGA Uniform Tumor数据集上提供了额外的结果,作为涵盖广泛组织类型、疾病、难度级别(最高108个类别)和染色(H&E和IHC)的代表性基准测试集。结果使用ABMIL和KNN(K=20)分别报告幻灯片和ROI任务。
请参阅UNI [1]和CONCH [2]论文以获取更详细的基准测试。
玻片基准测试
| 模型名称 | 预训练 | EBRAINS-C(12类,公开) | EBRAINS-F(30类,公开) | PANDA(5类,公开) | OncoTree-108(108类,内部) | IHC ER / PR 评估(6类,内部) |
|---|---|---|---|---|---|---|
| 平衡准确率 | 平衡准确率 | 二次加权 $\kappa$ | 平衡准确率 | 二次加权 $\kappa$ | ||
| UNI [1] | 视觉 | 0.883 | 0.675 | 0.946 | 0.538 | 0.785 |
| CONCH [2] | 视觉-语言 | 0.868 | 0.689 | 0.934 | 0.515 | 0.819 |
| Virchow (CLS+MEAN) [3] | 视觉 | 0.833 | 0.654 | 0.943 | 0.519 | 0.788 |
| Prov-GigaPath [4] | 视觉 | 0.875 | 0.687 | 0.942 | 0.522 | 0.821 |
| Phikon [5] | 视觉 | 0.810 | 0.659 | 0.950 | 0.486 | 0.744 |
| REMEDIS [6] | 视觉 | 0.687 | 0.382 | 0.932 | 0.412 | 0.762 |
| CTransPath [7] | 视觉 | 0.666 | 0.514 | 0.927 | 0.399 | 0.786 |
| Quilt-Net [8] | 视觉-语言 | 0.728 | 0.608 | 0.909 | 0.389 | 0.784 |
| PLIP [9] | 视觉-语言 | 0.683 | 0.562 | 0.901 | 0.369 | 0.759 |
| ResNet-50 (Tr) [10] | ImageNet 迁移 | 0.302 | 0.219 | 0.831 | 0.148 | 0.709 |
ROI 基准测试
| 模型名称 | 预训练 | CRC-100K-Raw (9 类,公开) | TCGA 统一肿瘤 (32 类,公开) |
|---|---|---|---|
| 平衡准确率 | 平衡准确率 | ||
| UNI [1] | 视觉 | 0.925 | 0.595 |
| CONCH [2] | 视觉-语言 | 0.941 | 0.556 |
| Virchow (CLS+MEAN) [3] | 视觉 | 0.919 | 0.549 |
| Virchow (CLS) [3] | 视觉 | 0.895 | 0.544 |
| Prov-GigaPath [4] | 视觉 | 0.929 | 0.593 |
| Phikon [5] | 视觉 | 0.845 | 0.533 |
| REMEDIS [6] | 视觉 | 0.908 | 0.541 |
| CTransPath [7] | 视觉 | 0.836 | 0.463 |
| Quilt-Net [8] | 视觉-语言 | 0.878 | 0.359 |
| PLIP [9] | 视觉-语言 | 0.840 | 0.370 |
| ResNet-50 [10] | ImageNet 迁移 | 0.797 | 0.318 |
许可和使用条款
ⓒ Mahmood 实验室。本模型及相关代码根据 CC-BY-NC-ND 4.0 许可发布,仅可用于非商业性学术研究目的,并需适当引用。禁止任何商业用途、销售或其他对 UNI 模型及其衍生品(包括使用 UNI 模型输出训练的模型或由 UNI 模型创建的数据集)的盈利行为,需事先获得批准。下载模型需要在 Hugging Face 上提前注册并同意使用条款。下载此模型即表示您同意不分发、发布或复制模型副本。如果您所在组织的其他用户希望使用 UNI 模型,他们必须作为个人用户注册并同意遵守使用条款。用户不得试图重新识别用于开发底层模型的去识别数据。如果您是商业实体,请联系通讯作者或 Mass General Brigham Innovation Office。
致谢
本项目基于诸如 ViT、DINOv2、LGSSL 和 Timm(ViT 模型实现)等优秀仓库构建。我们感谢这些作者和开发者的贡献。
参考文献
如果您在研究中发现我们的工作有用,或者使用了部分代码,请考虑引用我们的论文:
Chen, R.J., Ding, T., Lu, M.Y., Williamson, D.F.K., et al. Towards a general-purpose foundation model for computational pathology. Nat Med (2024). https://doi.org/10.1038/s41591-024-02857-3
@article{chen2024uni,
title={Towards a General-Purpose Foundation Model for Computational Pathology},
author={Chen, Richard J and Ding, Tong and Lu, Ming Y and Williamson, Drew FK and Jaume, Guillaume and Chen, Bowen and Zhang, Andrew and Shao, Daniel and Song, Andrew H and Shaban, Muhammad and others},
journal={Nature Medicine},
publisher={Nature Publishing Group},
year={2024}
}
<img src=.github/joint_logo.jpg>

 Github
Github Huggingface
Huggingface 论文
论文










